 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
SMILES 표기법을 사용하여 B형 간염 바이러스(HBV) 및 C형 간염 바이러스(HCV)에 대한 소분자의 항바이러스 활성을 예측하기 위해 새로운 계산 방법론인 HBCVTr이 개발되었습니다. 이 방법론은 양방향 및 자동 회귀 변환기(BART) 아키텍처를 원자별 및 분수별 토큰화 기술과 결합하여 SMILES의 순차적 정보와 화학적 특징을 포착합니다. 이 모델은 각각 HBV 및 HCV에 대한 1941개 및 7454개 화합물을 포함하는 엄선된 데이터세트에서 훈련 및 평가되었습니다. HBCVTr 모델은 높은 정확성과 강력한 예측 능력으로 다양한 머신러닝 모델과 기존 방법에 비해 우수한 성능을 달성했습니다. HBCVTr 모델을 사용한 가상 스크리닝을 통해 유망한 약동학적 특성을 지닌 잠재적인 HBV 및 HCV 억제제를 식별했습니다. 분자 도킹 및 분자 역학 시뮬레이션은 상위 후보 물질의 결합 친화도와 안정성을 더욱 검증하여 잠재적인 작용 메커니즘에 대한 통찰력을 제공합니다. 이 방법론은 HBV 및 HCV에 대한 새로운 항바이러스 치료법을 발견하고 설계하는 데 유용한 도구를 제공합니다.
Harnessing the Power of Artificial Intelligence for Drug Discovery: A Novel Virtual Screening Tool for Identifying Potential Inhibitors of Hepatitis B and C Viruses
신약 발견을 위한 인공 지능의 힘 활용: B형 및 C형 간염 바이러스의 잠재적 억제제를 식별하기 위한 새로운 가상 스크리닝 도구
Abstract
추상적인
Hepatitis B and C viruses (HBV and HCV) pose significant global health challenges, necessitating the development of novel and effective antiviral therapies. To accelerate this process, we present HBCVTr, an innovative virtual screening tool that leverages artificial intelligence (AI) to identify potential inhibitors against these viruses. Our methodology incorporates a bidirectional and auto-regressive transformer (BART) architecture, trained on a vast dataset of SMILES notations and biological activity data. The HBCVTr model demonstrates exceptional predictive performance, surpassing conventional machine learning approaches. Through virtual screening of a library of 10 million compounds, we identified promising candidates with favorable pharmacokinetic properties. Molecular docking and dynamics simulations confirmed the potential of these candidates as inhibitors of HBV and HCV. Our findings underscore the transformative potential of AI in drug discovery, offering a rapid and efficient approach to identify novel therapeutic options for combating viral infections.
B형 및 C형 간염 바이러스(HBV 및 HCV)는 전 세계적으로 심각한 건강 문제를 야기하므로 새롭고 효과적인 항바이러스 치료법의 개발이 필요합니다. 이 프로세스를 가속화하기 위해 우리는 인공 지능(AI)을 활용하여 이러한 바이러스에 대한 잠재적인 억제제를 식별하는 혁신적인 가상 검사 도구인 HBCVTr을 제시합니다. 우리의 방법론에는 방대한 SMILES 표기법 및 생물학적 활동 데이터 데이터 세트를 기반으로 훈련된 양방향 자동 회귀 변환기(BART) 아키텍처가 통합되어 있습니다. HBCVTr 모델은 기존의 기계 학습 접근 방식을 능가하는 탁월한 예측 성능을 보여줍니다. 1,000만 개 화합물 라이브러리의 가상 스크리닝을 통해 우리는 유리한 약동학적 특성을 가진 유망한 후보를 식별했습니다. 분자 도킹 및 역학 시뮬레이션을 통해 이러한 후보 물질이 HBV 및 HCV 억제제로서 잠재력을 확인했습니다. 우리의 연구 결과는 바이러스 감염 퇴치를 위한 새로운 치료 옵션을 식별하기 위한 신속하고 효율적인 접근 방식을 제공함으로써 신약 발견에서 AI의 혁신적인 잠재력을 강조합니다.
Introduction
소개
Hepatitis B and C viruses are prevalent pathogens that infect millions worldwide, leading to severe liver damage and potential life-threatening complications. Despite the availability of antiviral therapies, the need for new and improved treatments remains urgent, particularly in the face of emerging drug resistance. Traditional drug discovery processes are often time-consuming and expensive, prompting the exploration of alternative approaches.
B형 및 C형 간염 바이러스는 전 세계적으로 수백만 명을 감염시키는 만연한 병원체로, 심각한 간 손상과 잠재적으로 생명을 위협하는 합병증을 유발합니다. 항바이러스 치료법의 가용성에도 불구하고, 특히 새로운 약물 내성에 직면하여 새롭고 개선된 치료법의 필요성은 여전히 시급합니다. 전통적인 약물 발견 프로세스는 시간이 많이 걸리고 비용이 많이 들기 때문에 대체 접근법을 모색해야 합니다.
In this study, we present HBCVTr, a groundbreaking virtual screening tool that harnesses the power of AI to expedite the identification of potential HBV and HCV inhibitors. Our methodology utilizes a BART architecture, renowned for its ability to process sequential data, to predict the biological activity of small molecules using SMILES notations. This approach enables the rapid screening of vast chemical libraries, significantly accelerating the drug discovery process.
본 연구에서는 잠재적인 HBV 및 HCV 억제제 식별을 가속화하기 위해 AI의 힘을 활용하는 획기적인 가상 검사 도구인 HBCVTr을 제시합니다. 우리의 방법론은 순차 데이터 처리 능력으로 유명한 BART 아키텍처를 활용하여 SMILES 표기법을 사용하여 소분자의 생물학적 활동을 예측합니다. 이 접근 방식을 사용하면 방대한 화학 라이브러리를 신속하게 스크리닝할 수 있어 약물 발견 프로세스가 크게 가속화됩니다.
Methods
행동 양식
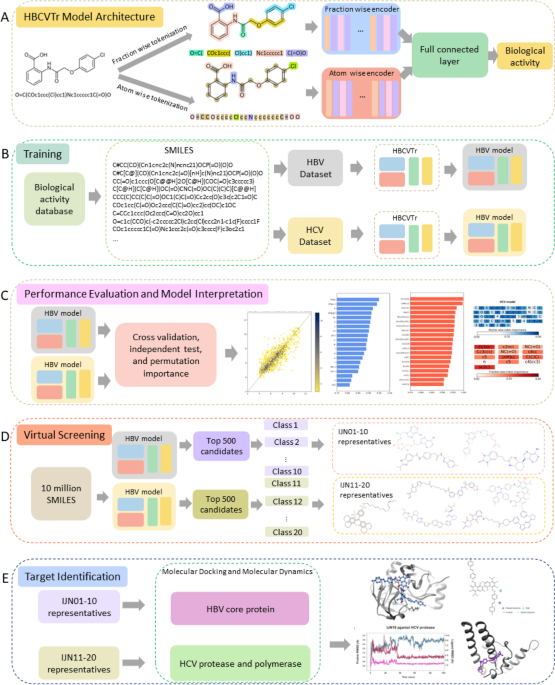
Data Collection and Preprocessing
데이터 수집 및 전처리
To train and evaluate our HBCVTr models, we curated antiviral activity assay data for HBV and HCV from the ChEMBL database. The data underwent rigorous filtering to ensure consistency and comparability, resulting in 1941 and 7454 compounds for HBV and HCV, respectively.
HBCVTr 모델을 훈련하고 평가하기 위해 ChEMBL 데이터베이스에서 HBV 및 HCV에 대한 항바이러스 활성 분석 데이터를 선별했습니다. 데이터는 일관성과 비교 가능성을 보장하기 위해 엄격한 필터링을 거쳐 HBV 및 HCV에 대해 각각 1941개 및 7454개의 화합물이 생성되었습니다.
SMILES notations, representing the molecular structures of the compounds, were preprocessed to remove salts and convert them into canonical SMILES using the RDKit package. These SMILES were subsequently tokenized into atom-wise and fraction-wise tokens, capturing both individual atoms and unique functional groups.
화합물의 분자 구조를 나타내는 SMILES 표기법은 RDKit 패키지를 사용하여 염을 제거하고 정식 SMILES로 변환하기 위해 전처리되었습니다. 이후 이러한 미소는 원자별 및 분수별 토큰으로 토큰화되어 개별 원자와 고유한 기능 그룹을 모두 포착했습니다.
Model Architecture and Training
모델 아키텍처 및 교육
Our HBCVTr model is based on a BART architecture, which is specifically designed for sequential data processing. The model comprises two encoders: one for atom-wise tokens and the other for fraction-wise tokens. These encoders leverage multi-head attention layers to learn the contextual relationships between tokens. The outputs from the encoders are concatenated and passed through fully connected layers, culminating in a regression head that predicts the biological activity of the input SMILES.
당사의 HBCVTr 모델은 순차 데이터 처리를 위해 특별히 설계된 BART 아키텍처를 기반으로 합니다. 모델은 두 개의 인코더로 구성됩니다. 하나는 원자 단위 토큰용이고 다른 하나는 분수 단위 토큰용입니다. 이러한 인코더는 멀티헤드 어텐션 레이어를 활용하여 토큰 간의 상황별 관계를 학습합니다. 인코더의 출력은 연결되어 완전히 연결된 레이어를 통과하여 입력 SMILES의 생물학적 활동을 예측하는 회귀 헤드로 끝납니다.
We optimized the model's hyperparameters through a comprehensive grid search, ensuring optimal performance. The model was trained on 72% of the data, while 8% and 20% were allocated for validation and independent testing, respectively.
포괄적인 그리드 검색을 통해 모델의 하이퍼파라미터를 최적화하여 최적의 성능을 보장했습니다. 모델은 데이터의 72%에 대해 학습되었으며, 검증 및 독립적 테스트에 각각 8%와 20%가 할당되었습니다.
Evaluation Criteria
평가 기준
To assess the predictive performance of HBCVTr, we employed a suite of regression evaluation metrics, including mean square error (MSE), mean absolute error (MAE), root mean square error (RMSE), R-squared, Pearson's correlation coefficient (PCC), and Spearman rank correlation (Spearman). These metrics evaluate the model's ability to accurately predict biological activity values.
HBCVTr의 예측 성능을 평가하기 위해 평균 제곱 오차(MSE), 평균 절대 오차(MAE), 평균 제곱근 오차(RMSE), R-제곱, 피어슨 상관 계수(PCC)를 포함한 일련의 회귀 평가 측정 항목을 사용했습니다. 및 Spearman 순위 상관관계(Spearman). 이러한 측정항목은 생물학적 활동 값을 정확하게 예측하는 모델의 능력을 평가합니다.
Virtual Screening and Pharmacokinetic Properties Prediction
가상 스크리닝 및 약동학적 특성 예측
The trained HBCVTr models were utilized for virtual screening of a library of 10 million compounds. The top candidates with the highest predicted biological activity were further evaluated for their pharmacokinetic properties using the SwissADME web tool. This assessment ensured the identification of compounds with desirable drug-like characteristics.
훈련된 HBCVTr 모델은 천만 개의 화합물 라이브러리의 가상 스크리닝에 활용되었습니다. 예측된 생물학적 활성이 가장 높은 상위 후보 물질은 SwissADME 웹 도구를 사용하여 약동학적 특성에 대해 추가로 평가되었습니다. 이 평가를 통해 바람직한 약물 유사 특성을 가진 화합물의 식별이 보장되었습니다.
Molecular Docking and Molecular Dynamics Simulation
분자 도킹 및 분자 역학 시뮬레이션
To investigate the potential binding of the top candidates to target proteins, molecular docking was performed using Autodock Vina. The stability of the protein-ligand complexes was subsequently assessed through molecular dynamics simulations using the Desmond Molecular Dynamics System. These simulations provided insights into the interactions between the candidates and their target proteins.
표적 단백질에 대한 상위 후보의 잠재적 결합을 조사하기 위해 Autodock Vina를 사용하여 분자 도킹을 수행했습니다. 단백질-리간드 복합체의 안정성은 이후 Desmond Molecular Dynamics System을 사용한 분자 역학 시뮬레이션을 통해 평가되었습니다. 이러한 시뮬레이션은 후보 단백질과 표적 단백질 간의 상호 작용에 대한 통찰력을 제공했습니다.
Results
결과
Model Performance
모델 성능
The HBCVTr models demonstrated remarkable predictive performance on both HBV and HCV datasets. They outperformed conventional machine learning approaches, consistently achieving higher R-squared and PCC values. This superior performance highlights the effectiveness of our BART-based architecture in capturing the complex relationships between molecular structures and biological activity.
HBCVTr 모델은 HBV 및 HCV 데이터 세트 모두에서 놀라운 예측 성능을 보여주었습니다. 그들은 기존의 기계 학습 접근 방식을 능가하여 지속적으로 더 높은 R-제곱 및 PCC 값을 달성했습니다. 이 뛰어난 성능은 분자 구조와 생물학적 활동 사이의 복잡한 관계를 포착하는 데 있어 BART 기반 아키텍처의 효율성을 강조합니다.
Virtual Screening and Pharmacokinetic Properties
가상 스크리닝 및 약동학적 특성
Virtual screening of 10 million compounds identified promising candidates with high predicted biological activity against HBV and HCV. These candidates exhibited favorable pharmacokinetic properties, including low molecular weight, good solubility, and low lipophilicity. Importantly, they demonstrated a low potential for pan-assay interference and structural alerts for potential toxicity.
1,000만 개의 화합물에 대한 가상 스크리닝을 통해 HBV 및 HCV에 대해 생물학적 활성이 높게 예측되는 유망한 후보를 식별했습니다. 이들 후보물질은 저분자량, 우수한 용해도, 낮은 친지성 등 유리한 약동학적 특성을 나타냈습니다. 중요한 것은 범 분석 간섭 가능성이 낮고 잠재적 독성에 대한 구조적 경고가 있음을 입증했다는 것입니다.
Molecular Docking and Molecular Dynamics Simulation
분자 도킹 및 분자 역학 시뮬레이션
Molecular docking and molecular dynamics simulations revealed the potential binding modes of the top candidates to target proteins. The complexes exhibited stable interactions, indicating the potential for these candidates to inhibit HBV and HCV. Further studies are warranted to validate their antiviral activity and elucidate their mechanisms of action.
분자 도킹 및 분자 역학 시뮬레이션을 통해 표적 단백질에 대한 상위 후보의 잠재적 결합 모드가 밝혀졌습니다. 복합체는 안정적인 상호작용을 나타냈는데, 이는 이들 후보물질이 HBV 및 HCV를 억제할 가능성이 있음을 나타냅니다. 항바이러스 활성을 검증하고 작용 메커니즘을 밝히기 위해서는 추가 연구가 필요합니다.
Discussion
논의
The HBCVTr virtual screening tool represents a significant advancement in drug discovery for HBV and HCV. Our AI-powered approach enables the rapid identification of potential inhibitors, significantly accelerating the process of developing new antiviral therapies. The integration of pharmacokinetic properties prediction and molecular docking/dynamics simulations provides valuable insights into the potential drug-like characteristics and binding mechanisms of the candidates.
HBCVTr 가상 스크리닝 도구는 HBV 및 HCV에 대한 약물 발견에 있어 상당한 발전을 나타냅니다. 당사의 AI 기반 접근 방식을 통해 잠재적인 억제제를 신속하게 식별하여 새로운 항바이러스 치료법 개발 과정을 크게 가속화할 수 있습니다. 약동학적 특성 예측과 분자 도킹/역학 시뮬레이션의 통합은 후보 물질의 잠재적인 약물 유사 특성 및 결합 메커니즘에 대한 귀중한 통찰력을 제공합니다.
The HBCVTr tool has broad implications for the field of drug discovery. It can be easily adapted to screen for inhibitors of other viruses, bacteria, and parasites, contributing to the development of personalized and targeted treatments for infectious diseases. Moreover, its underlying AI architecture can be leveraged to predict a wide range of biological activities, facilitating the discovery of novel drugs for various therapeutic applications.
HBCVTr 도구는 약물 발견 분야에 광범위한 영향을 미칩니다. 다른 바이러스, 박테리아 및 기생충의 억제제를 검사하는 데 쉽게 적용할 수 있어 전염병에 대한 맞춤형 및 표적 치료법 개발에 기여합니다. 또한 기본 AI 아키텍처를 활용하여 광범위한 생물학적 활동을 예측하여 다양한 치료 응용 분야를 위한 새로운 약물의 발견을 촉진할 수 있습니다.
Conclusion
결론
In conclusion, the HBCVTr virtual screening tool is a powerful AI-driven platform that transforms the drug discovery process for HBV and HCV. Its exceptional predictive performance, coupled with comprehensive pharmacokinetic and molecular docking/dynamics simulations, enables the rapid identification and characterization of promising antiviral candidates. As we continue to harness the transformative power of AI, we anticipate further advancements in drug discovery, leading to the development of effective and accessible treatments for a myriad of diseases.
결론적으로, HBCVTr 가상 스크리닝 도구는 HBV 및 HCV에 대한 약물 발견 프로세스를 변화시키는 강력한 AI 기반 플랫폼입니다. 포괄적인 약동학 및 분자 도킹/역학 시뮬레이션과 결합된 탁월한 예측 성능을 통해 유망한 항바이러스 후보의 신속한 식별 및 특성화를 가능하게 합니다. 우리는 AI의 혁신적인 힘을 계속 활용하면서 신약 발견이 더욱 발전하여 수많은 질병에 대한 효과적이고 접근 가능한 치료법이 개발될 것으로 기대합니다.
부인 성명:info@kdj.com
제공된 정보는 거래 조언이 아닙니다. kdj.com은 이 기사에 제공된 정보를 기반으로 이루어진 투자에 대해 어떠한 책임도 지지 않습니다. 암호화폐는 변동성이 매우 높으므로 철저한 조사 후 신중하게 투자하는 것이 좋습니다!
본 웹사이트에 사용된 내용이 귀하의 저작권을 침해한다고 판단되는 경우, 즉시 당사(info@kdj.com)로 연락주시면 즉시 삭제하도록 하겠습니다.
-

-

-

-

-

-

- 중국은 북동쪽에서 1,000 톤의 금 보증금을 발견했습니다
- 2025-04-03 10:15:21
- 지질 학자들은 중국 북동부에서 1,000 톤 금 예금이 발견되었다고 발표했다.
-

-

-






























































