 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
新しい計算手法である HBCVTr は、SMILES 表記を使用して B 型肝炎ウイルス (HBV) および C 型肝炎ウイルス (HCV) に対する小分子の抗ウイルス活性を予測するために開発されました。この方法論は、双方向自己回帰変換器 (BART) アーキテクチャと原子単位および分数単位のトークン化技術を組み合わせて、SMILES の逐次情報と化学的特徴を捕捉します。モデルは、HBV と HCV それぞれについて 1941 と 7454 の化合物を含む精選されたデータセットでトレーニングおよび評価されました。 HBCVTr モデルは、さまざまな機械学習モデルや既存の手法と比較して優れたパフォーマンスを実現し、高精度で堅牢な予測機能を備えています。 HBCVTr モデルを使用した仮想スクリーニングにより、有望な薬物動態特性を持つ潜在的な HBV および HCV 阻害剤が同定されました。分子ドッキングと分子動力学シミュレーションにより、上位候補の結合親和性と安定性がさらに検証され、それらの潜在的な作用機序についての洞察が得られました。この方法論は、HBV および HCV に対する新しい抗ウイルス療法を発見および設計するための貴重なツールを提供します。
Harnessing the Power of Artificial Intelligence for Drug Discovery: A Novel Virtual Screening Tool for Identifying Potential Inhibitors of Hepatitis B and C Viruses
人工知能の力を創薬に活用: B 型および C 型肝炎ウイルスの潜在的な阻害剤を特定するための新しい仮想スクリーニング ツール
Abstract
抽象的な
Hepatitis B and C viruses (HBV and HCV) pose significant global health challenges, necessitating the development of novel and effective antiviral therapies. To accelerate this process, we present HBCVTr, an innovative virtual screening tool that leverages artificial intelligence (AI) to identify potential inhibitors against these viruses. Our methodology incorporates a bidirectional and auto-regressive transformer (BART) architecture, trained on a vast dataset of SMILES notations and biological activity data. The HBCVTr model demonstrates exceptional predictive performance, surpassing conventional machine learning approaches. Through virtual screening of a library of 10 million compounds, we identified promising candidates with favorable pharmacokinetic properties. Molecular docking and dynamics simulations confirmed the potential of these candidates as inhibitors of HBV and HCV. Our findings underscore the transformative potential of AI in drug discovery, offering a rapid and efficient approach to identify novel therapeutic options for combating viral infections.
B 型および C 型肝炎ウイルス (HBV および HCV) は、世界的な健康上の重大な課題を引き起こしており、新規で効果的な抗ウイルス療法の開発が必要です。このプロセスを加速するために、人工知能 (AI) を活用してこれらのウイルスに対する潜在的な阻害剤を特定する革新的な仮想スクリーニング ツールである HBCVTr を紹介します。私たちの方法論には、SMILES 表記法と生物活性データの膨大なデータセットでトレーニングされた双方向自己回帰変換器 (BART) アーキテクチャが組み込まれています。 HBCVTr モデルは、従来の機械学習アプローチを上回る、優れた予測パフォーマンスを示します。 1,000 万の化合物ライブラリの仮想スクリーニングを通じて、好ましい薬物動態特性を持つ有望な候補を特定しました。分子ドッキングおよび動力学シミュレーションにより、これらの候補がHBVおよびHCVの阻害剤としての可能性を確認した。私たちの調査結果は、創薬における AI の変革の可能性を強調し、ウイルス感染と戦うための新しい治療選択肢を特定するための迅速かつ効率的なアプローチを提供します。
Introduction
導入
Hepatitis B and C viruses are prevalent pathogens that infect millions worldwide, leading to severe liver damage and potential life-threatening complications. Despite the availability of antiviral therapies, the need for new and improved treatments remains urgent, particularly in the face of emerging drug resistance. Traditional drug discovery processes are often time-consuming and expensive, prompting the exploration of alternative approaches.
B 型および C 型肝炎ウイルスは、世界中で何百万人もの人が感染する流行病原体であり、重篤な肝障害や生命を脅かす合併症を引き起こす可能性があります。抗ウイルス療法が利用可能になったにもかかわらず、特に薬剤耐性の出現に直面して、新しく改良された治療法の必要性は依然として緊急である。従来の創薬プロセスは時間と費用がかかることが多く、代替アプローチの模索が求められています。
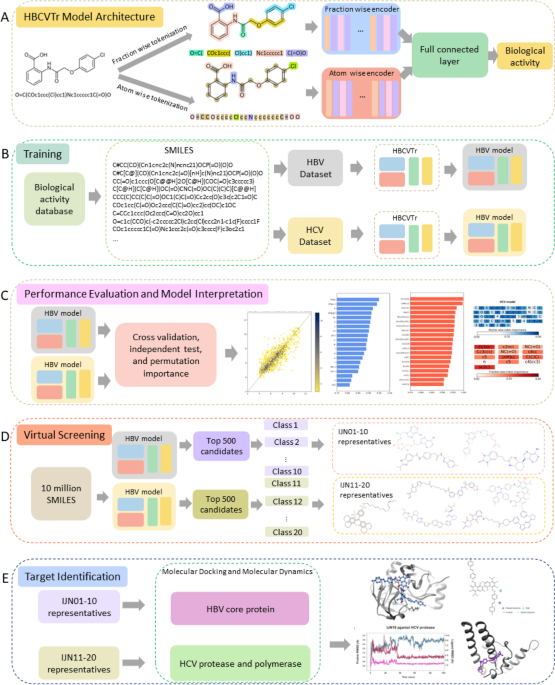
In this study, we present HBCVTr, a groundbreaking virtual screening tool that harnesses the power of AI to expedite the identification of potential HBV and HCV inhibitors. Our methodology utilizes a BART architecture, renowned for its ability to process sequential data, to predict the biological activity of small molecules using SMILES notations. This approach enables the rapid screening of vast chemical libraries, significantly accelerating the drug discovery process.
この研究では、AI の力を利用して潜在的な HBV および HCV 阻害剤の特定を迅速化する画期的な仮想スクリーニング ツール HBCVTr を紹介します。私たちの方法論は、連続データを処理する能力で有名な BART アーキテクチャを利用し、SMILES 表記を使用して小分子の生物学的活性を予測します。このアプローチにより、膨大な化学ライブラリの迅速なスクリーニングが可能になり、創薬プロセスが大幅に加速されます。
Methods
メソッド
Data Collection and Preprocessing
データの収集と前処理
To train and evaluate our HBCVTr models, we curated antiviral activity assay data for HBV and HCV from the ChEMBL database. The data underwent rigorous filtering to ensure consistency and comparability, resulting in 1941 and 7454 compounds for HBV and HCV, respectively.
HBCVTr モデルをトレーニングおよび評価するために、ChEMBL データベースから HBV および HCV の抗ウイルス活性アッセイ データを厳選しました。データは一貫性と比較可能性を確保するために厳格なフィルタリングを受け、その結果、HBV と HCV についてそれぞれ 1941 と 7454 の化合物が得られました。
SMILES notations, representing the molecular structures of the compounds, were preprocessed to remove salts and convert them into canonical SMILES using the RDKit package. These SMILES were subsequently tokenized into atom-wise and fraction-wise tokens, capturing both individual atoms and unique functional groups.
化合物の分子構造を表す SMILES 表記は、RDKit パッケージを使用して塩を除去し、正規の SMILES に変換するために前処理されました。これらの SMILE はその後、原子ごとおよび分数ごとのトークンにトークン化され、個々の原子と固有の官能基の両方が捕捉されました。
Model Architecture and Training
モデルのアーキテクチャとトレーニング
Our HBCVTr model is based on a BART architecture, which is specifically designed for sequential data processing. The model comprises two encoders: one for atom-wise tokens and the other for fraction-wise tokens. These encoders leverage multi-head attention layers to learn the contextual relationships between tokens. The outputs from the encoders are concatenated and passed through fully connected layers, culminating in a regression head that predicts the biological activity of the input SMILES.
当社の HBCVTr モデルは、特にシーケンシャル データ処理用に設計された BART アーキテクチャに基づいています。モデルは 2 つのエンコーダーで構成されます。1 つはアトム単位のトークン用で、もう 1 つは分数単位のトークン用です。これらのエンコーダーは、マルチヘッド アテンション レイヤーを活用して、トークン間の文脈上の関係を学習します。エンコーダーからの出力は連結され、完全に接続された層を通過し、最終的に入力 SMILES の生物活性を予測する回帰ヘッドに到達します。
We optimized the model's hyperparameters through a comprehensive grid search, ensuring optimal performance. The model was trained on 72% of the data, while 8% and 20% were allocated for validation and independent testing, respectively.
包括的なグリッド検索を通じてモデルのハイパーパラメーターを最適化し、最適なパフォーマンスを確保しました。モデルはデータの 72% でトレーニングされ、8% と 20% がそれぞれ検証と独立したテストに割り当てられました。
Evaluation Criteria
評価基準
To assess the predictive performance of HBCVTr, we employed a suite of regression evaluation metrics, including mean square error (MSE), mean absolute error (MAE), root mean square error (RMSE), R-squared, Pearson's correlation coefficient (PCC), and Spearman rank correlation (Spearman). These metrics evaluate the model's ability to accurately predict biological activity values.
HBCVTr の予測パフォーマンスを評価するために、平均二乗誤差 (MSE)、平均絶対誤差 (MAE)、二乗平均平方根誤差 (RMSE)、R 二乗、ピアソン相関係数 (PCC) を含む一連の回帰評価指標を採用しました。 、およびスピアマンのランク相関 (スピアマン)。これらのメトリクスは、生物活性値を正確に予測するモデルの能力を評価します。
Virtual Screening and Pharmacokinetic Properties Prediction
仮想スクリーニングと薬物動態特性の予測
The trained HBCVTr models were utilized for virtual screening of a library of 10 million compounds. The top candidates with the highest predicted biological activity were further evaluated for their pharmacokinetic properties using the SwissADME web tool. This assessment ensured the identification of compounds with desirable drug-like characteristics.
トレーニングされた HBCVTr モデルは、1,000 万の化合物ライブラリの仮想スクリーニングに利用されました。最も高い予測生物学的活性を持つ上位候補は、SwissADME Web ツールを使用して薬物動態特性についてさらに評価されました。この評価により、望ましい薬物のような特性を持つ化合物の同定が確実に行われました。
Molecular Docking and Molecular Dynamics Simulation
分子ドッキングと分子動力学シミュレーション
To investigate the potential binding of the top candidates to target proteins, molecular docking was performed using Autodock Vina. The stability of the protein-ligand complexes was subsequently assessed through molecular dynamics simulations using the Desmond Molecular Dynamics System. These simulations provided insights into the interactions between the candidates and their target proteins.
上位候補の標的タンパク質への潜在的な結合を調査するために、Autodock Vina を使用して分子ドッキングを実行しました。その後、タンパク質-リガンド複合体の安定性は、Desmond Molecular Dynamics System を使用した分子動力学シミュレーションによって評価されました。これらのシミュレーションにより、候補とその標的タンパク質の間の相互作用についての洞察が得られました。
Results
結果
Model Performance
モデルのパフォーマンス
The HBCVTr models demonstrated remarkable predictive performance on both HBV and HCV datasets. They outperformed conventional machine learning approaches, consistently achieving higher R-squared and PCC values. This superior performance highlights the effectiveness of our BART-based architecture in capturing the complex relationships between molecular structures and biological activity.
HBCVTr モデルは、HBV データセットと HCV データセットの両方で顕著な予測パフォーマンスを実証しました。これらは従来の機械学習アプローチを上回り、一貫してより高い R 二乗値と PCC 値を達成しました。この優れたパフォーマンスは、分子構造と生物学的活性の間の複雑な関係を捕捉する際の当社の BART ベースのアーキテクチャの有効性を強調しています。
Virtual Screening and Pharmacokinetic Properties
仮想スクリーニングと薬物動態特性
Virtual screening of 10 million compounds identified promising candidates with high predicted biological activity against HBV and HCV. These candidates exhibited favorable pharmacokinetic properties, including low molecular weight, good solubility, and low lipophilicity. Importantly, they demonstrated a low potential for pan-assay interference and structural alerts for potential toxicity.
1,000 万の化合物の仮想スクリーニングにより、HBV および HCV に対して高い生物学的活性が予測される有望な候補が特定されました。これらの候補は、低分子量、良好な溶解性、低親油性などの好ましい薬物動態特性を示しました。重要なのは、それらは汎アッセイ干渉の可能性が低いことと、潜在的な毒性に対する構造的警告を示したことです。
Molecular Docking and Molecular Dynamics Simulation
分子ドッキングと分子動力学シミュレーション
Molecular docking and molecular dynamics simulations revealed the potential binding modes of the top candidates to target proteins. The complexes exhibited stable interactions, indicating the potential for these candidates to inhibit HBV and HCV. Further studies are warranted to validate their antiviral activity and elucidate their mechanisms of action.
分子ドッキングと分子動力学シミュレーションにより、上位候補の標的タンパク質への潜在的な結合モードが明らかになりました。複合体は安定した相互作用を示し、これらの候補が HBV および HCV を阻害する可能性があることを示しました。それらの抗ウイルス活性を検証し、作用機序を解明するには、さらなる研究が必要です。
Discussion
議論
The HBCVTr virtual screening tool represents a significant advancement in drug discovery for HBV and HCV. Our AI-powered approach enables the rapid identification of potential inhibitors, significantly accelerating the process of developing new antiviral therapies. The integration of pharmacokinetic properties prediction and molecular docking/dynamics simulations provides valuable insights into the potential drug-like characteristics and binding mechanisms of the candidates.
HBCVTr 仮想スクリーニング ツールは、HBV および HCV の創薬において大きな進歩をもたらします。当社の AI を活用したアプローチにより、潜在的な阻害剤を迅速に特定できるようになり、新しい抗ウイルス療法の開発プロセスが大幅に加速されます。薬物動態学的特性の予測と分子ドッキング/動力学シミュレーションの統合により、候補物質の潜在的な薬物様特性と結合機構についての貴重な洞察が得られます。
The HBCVTr tool has broad implications for the field of drug discovery. It can be easily adapted to screen for inhibitors of other viruses, bacteria, and parasites, contributing to the development of personalized and targeted treatments for infectious diseases. Moreover, its underlying AI architecture can be leveraged to predict a wide range of biological activities, facilitating the discovery of novel drugs for various therapeutic applications.
HBCVTr ツールは、創薬の分野に広範な影響を及ぼします。他のウイルス、細菌、寄生虫の阻害剤のスクリーニングに簡単に適応でき、感染症に対する個別化された標的治療法の開発に貢献します。さらに、その基盤となる AI アーキテクチャを活用して、幅広い生物学的活動を予測することができ、さまざまな治療用途のための新薬の発見を促進します。
Conclusion
結論
In conclusion, the HBCVTr virtual screening tool is a powerful AI-driven platform that transforms the drug discovery process for HBV and HCV. Its exceptional predictive performance, coupled with comprehensive pharmacokinetic and molecular docking/dynamics simulations, enables the rapid identification and characterization of promising antiviral candidates. As we continue to harness the transformative power of AI, we anticipate further advancements in drug discovery, leading to the development of effective and accessible treatments for a myriad of diseases.
結論として、HBCVTr 仮想スクリーニング ツールは、HBV および HCV の創薬プロセスを変革する強力な AI 駆動プラットフォームです。その卓越した予測性能と包括的な薬物動態および分子ドッキング/ダイナミクス シミュレーションを組み合わせることで、有望な抗ウイルス薬候補の迅速な同定と特性評価が可能になります。 AI の変革力を活用し続けることで、創薬がさらに進歩し、無数の疾患に対する効果的で利用可能な治療法の開発につながることが期待されます。
免責事項:info@kdj.com
提供される情報は取引に関するアドバイスではありません。 kdj.com は、この記事で提供される情報に基づいて行われた投資に対して一切の責任を負いません。暗号通貨は変動性が高いため、十分な調査を行った上で慎重に投資することを強くお勧めします。
このウェブサイトで使用されているコンテンツが著作権を侵害していると思われる場合は、直ちに当社 (info@kdj.com) までご連絡ください。速やかに削除させていただきます。




















![取引は、[ビデオのレビュー]ゴールドビットコイン原油注文に従うことです。 取引は、[ビデオのレビュー]ゴールドビットコイン原油注文に従うことです。](/uploads/2025/04/26/cryptocurrencies-news/videos/trading-follow-review-video-gold-bitcoin-crude-oil-profits/image-1.webp)





































