 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
Articles d’actualité sur les crypto-monnaies
Exploiter l'intelligence artificielle pour lutter contre l'hépatite : un outil de dépistage virtuel identifie les inhibiteurs dormants
Apr 23, 2024 at 05:02 am
Une nouvelle méthodologie informatique, HBCVTr, a été développée pour prédire l'activité antivirale de petites molécules contre le virus de l'hépatite B (VHB) et le virus de l'hépatite C (VHC) à l'aide de leurs notations SMILES. Cette méthodologie combine une architecture de transformateur bidirectionnel et auto-régressif (BART) avec des techniques de tokenisation par atomes et par fractions pour capturer les informations séquentielles et les caractéristiques chimiques de SMILES. Les modèles ont été formés et évalués sur des ensembles de données sélectionnés contenant respectivement 1 941 et 7 454 composés pour le VHB et le VHC. Les modèles HBCVTr ont atteint des performances supérieures à celles de divers modèles d'apprentissage automatique et méthodes existantes, avec une grande précision et des capacités de prédiction robustes. Le criblage virtuel utilisant les modèles HBCVTr a identifié des inhibiteurs potentiels du VHB et du VHC dotés de propriétés pharmacocinétiques prometteuses. Les simulations d’amarrage moléculaire et de dynamique moléculaire ont en outre validé l’affinité de liaison et la stabilité des meilleurs candidats, fournissant ainsi un aperçu de leurs mécanismes d’action potentiels. Cette méthodologie offre un outil précieux pour découvrir et concevoir de nouvelles thérapies antivirales contre le VHB et le VHC.
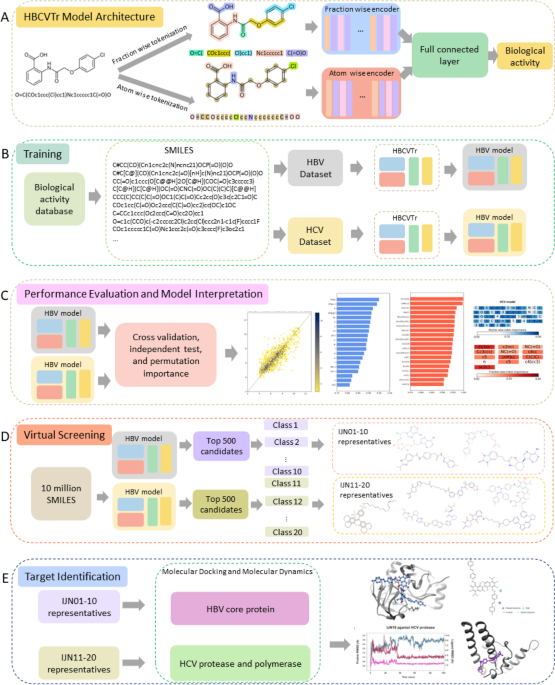
Harnessing the Power of Artificial Intelligence for Drug Discovery: A Novel Virtual Screening Tool for Identifying Potential Inhibitors of Hepatitis B and C Viruses
Exploiter la puissance de l'intelligence artificielle pour la découverte de médicaments : un nouvel outil de dépistage virtuel pour identifier les inhibiteurs potentiels des virus de l'hépatite B et C
Abstract
Abstrait
Hepatitis B and C viruses (HBV and HCV) pose significant global health challenges, necessitating the development of novel and effective antiviral therapies. To accelerate this process, we present HBCVTr, an innovative virtual screening tool that leverages artificial intelligence (AI) to identify potential inhibitors against these viruses. Our methodology incorporates a bidirectional and auto-regressive transformer (BART) architecture, trained on a vast dataset of SMILES notations and biological activity data. The HBCVTr model demonstrates exceptional predictive performance, surpassing conventional machine learning approaches. Through virtual screening of a library of 10 million compounds, we identified promising candidates with favorable pharmacokinetic properties. Molecular docking and dynamics simulations confirmed the potential of these candidates as inhibitors of HBV and HCV. Our findings underscore the transformative potential of AI in drug discovery, offering a rapid and efficient approach to identify novel therapeutic options for combating viral infections.
Les virus de l’hépatite B et C (VHB et VHC) posent d’importants défis de santé mondiaux, nécessitant le développement de thérapies antivirales nouvelles et efficaces. Pour accélérer ce processus, nous présentons HBCVTr, un outil de dépistage virtuel innovant qui exploite l’intelligence artificielle (IA) pour identifier les inhibiteurs potentiels de ces virus. Notre méthodologie intègre une architecture de transformateur bidirectionnel et auto-régressif (BART), formée sur un vaste ensemble de données de notations SMILES et de données d'activité biologique. Le modèle HBCVTr démontre des performances prédictives exceptionnelles, surpassant les approches conventionnelles d’apprentissage automatique. Grâce au criblage virtuel d’une bibliothèque de 10 millions de composés, nous avons identifié des candidats prometteurs dotés de propriétés pharmacocinétiques favorables. Les simulations moléculaires et dynamiques ont confirmé le potentiel de ces candidats en tant qu’inhibiteurs du VHB et du VHC. Nos résultats soulignent le potentiel transformateur de l’IA dans la découverte de médicaments, offrant une approche rapide et efficace pour identifier de nouvelles options thérapeutiques pour lutter contre les infections virales.
Introduction
Introduction
Hepatitis B and C viruses are prevalent pathogens that infect millions worldwide, leading to severe liver damage and potential life-threatening complications. Despite the availability of antiviral therapies, the need for new and improved treatments remains urgent, particularly in the face of emerging drug resistance. Traditional drug discovery processes are often time-consuming and expensive, prompting the exploration of alternative approaches.
Les virus de l’hépatite B et C sont des agents pathogènes répandus qui infectent des millions de personnes dans le monde, entraînant de graves lésions hépatiques et des complications potentiellement mortelles. Malgré la disponibilité de thérapies antivirales, le besoin de traitements nouveaux et améliorés reste urgent, en particulier face à l’émergence d’une résistance aux médicaments. Les processus traditionnels de découverte de médicaments prennent souvent du temps et sont coûteux, ce qui incite à explorer des approches alternatives.
In this study, we present HBCVTr, a groundbreaking virtual screening tool that harnesses the power of AI to expedite the identification of potential HBV and HCV inhibitors. Our methodology utilizes a BART architecture, renowned for its ability to process sequential data, to predict the biological activity of small molecules using SMILES notations. This approach enables the rapid screening of vast chemical libraries, significantly accelerating the drug discovery process.
Dans cette étude, nous présentons HBCVTr, un outil de dépistage virtuel révolutionnaire qui exploite la puissance de l’IA pour accélérer l’identification des inhibiteurs potentiels du VHB et du VHC. Notre méthodologie utilise une architecture BART, réputée pour sa capacité à traiter des données séquentielles, pour prédire l'activité biologique de petites molécules à l'aide des notations SMILES. Cette approche permet le criblage rapide de vastes bibliothèques chimiques, accélérant ainsi considérablement le processus de découverte de médicaments.
Methods
Méthodes
Data Collection and Preprocessing
Collecte et prétraitement des données
To train and evaluate our HBCVTr models, we curated antiviral activity assay data for HBV and HCV from the ChEMBL database. The data underwent rigorous filtering to ensure consistency and comparability, resulting in 1941 and 7454 compounds for HBV and HCV, respectively.
Pour former et évaluer nos modèles HBCVTr, nous avons rassemblé les données de test d'activité antivirale pour le VHB et le VHC à partir de la base de données ChEMBL. Les données ont été soumises à un filtrage rigoureux pour garantir la cohérence et la comparabilité, ce qui a abouti à 1 941 et 7 454 composés pour le VHB et le VHC, respectivement.
SMILES notations, representing the molecular structures of the compounds, were preprocessed to remove salts and convert them into canonical SMILES using the RDKit package. These SMILES were subsequently tokenized into atom-wise and fraction-wise tokens, capturing both individual atoms and unique functional groups.
Les notations SMILES, représentant les structures moléculaires des composés, ont été prétraitées pour éliminer les sels et les convertir en SMILES canoniques à l'aide du package RDKit. Ces SMILES ont ensuite été symbolisés en jetons atomiques et fractionnaires, capturant à la fois des atomes individuels et des groupes fonctionnels uniques.
Model Architecture and Training
Architecture du modèle et formation
Our HBCVTr model is based on a BART architecture, which is specifically designed for sequential data processing. The model comprises two encoders: one for atom-wise tokens and the other for fraction-wise tokens. These encoders leverage multi-head attention layers to learn the contextual relationships between tokens. The outputs from the encoders are concatenated and passed through fully connected layers, culminating in a regression head that predicts the biological activity of the input SMILES.
Notre modèle HBCVTr est basé sur une architecture BART, spécifiquement conçue pour le traitement séquentiel de données. Le modèle comprend deux encodeurs : un pour les jetons atomiques et l'autre pour les jetons fractionnaires. Ces encodeurs exploitent les couches d'attention multi-têtes pour apprendre les relations contextuelles entre les jetons. Les sorties des encodeurs sont concaténées et transmises à travers des couches entièrement connectées, aboutissant à une tête de régression qui prédit l'activité biologique des SMILES d'entrée.
We optimized the model's hyperparameters through a comprehensive grid search, ensuring optimal performance. The model was trained on 72% of the data, while 8% and 20% were allocated for validation and independent testing, respectively.
Nous avons optimisé les hyperparamètres du modèle grâce à une recherche de grille complète, garantissant des performances optimales. Le modèle a été formé sur 72 % des données, tandis que 8 % et 20 % ont été alloués respectivement à la validation et aux tests indépendants.
Evaluation Criteria
Critère d'évaluation
To assess the predictive performance of HBCVTr, we employed a suite of regression evaluation metrics, including mean square error (MSE), mean absolute error (MAE), root mean square error (RMSE), R-squared, Pearson's correlation coefficient (PCC), and Spearman rank correlation (Spearman). These metrics evaluate the model's ability to accurately predict biological activity values.
Pour évaluer les performances prédictives de HBCVTr, nous avons utilisé une suite de mesures d'évaluation de régression, notamment l'erreur quadratique moyenne (MSE), l'erreur absolue moyenne (MAE), l'erreur quadratique moyenne (RMSE), le R carré et le coefficient de corrélation de Pearson (PCC). et corrélation de rang de Spearman (Spearman). Ces mesures évaluent la capacité du modèle à prédire avec précision les valeurs de l'activité biologique.
Virtual Screening and Pharmacokinetic Properties Prediction
Criblage virtuel et prédiction des propriétés pharmacocinétiques
The trained HBCVTr models were utilized for virtual screening of a library of 10 million compounds. The top candidates with the highest predicted biological activity were further evaluated for their pharmacokinetic properties using the SwissADME web tool. This assessment ensured the identification of compounds with desirable drug-like characteristics.
Les modèles HBCVTr formés ont été utilisés pour le criblage virtuel d’une bibliothèque de 10 millions de composés. Les meilleurs candidats ayant l'activité biologique prévue la plus élevée ont ensuite été évalués pour leurs propriétés pharmacocinétiques à l'aide de l'outil Web SwissADME. Cette évaluation a permis d’identifier des composés présentant des caractéristiques souhaitables de type médicament.
Molecular Docking and Molecular Dynamics Simulation
Docking moléculaire et simulation de dynamique moléculaire
To investigate the potential binding of the top candidates to target proteins, molecular docking was performed using Autodock Vina. The stability of the protein-ligand complexes was subsequently assessed through molecular dynamics simulations using the Desmond Molecular Dynamics System. These simulations provided insights into the interactions between the candidates and their target proteins.
Pour étudier la liaison potentielle des meilleurs candidats aux protéines cibles, un amarrage moléculaire a été réalisé à l’aide d’Autodock Vina. La stabilité des complexes protéine-ligand a ensuite été évaluée au moyen de simulations de dynamique moléculaire à l’aide du système Desmond Molecular Dynamics. Ces simulations ont fourni des informations sur les interactions entre les candidats et leurs protéines cibles.
Results
Résultats
Model Performance
Performances du modèle
The HBCVTr models demonstrated remarkable predictive performance on both HBV and HCV datasets. They outperformed conventional machine learning approaches, consistently achieving higher R-squared and PCC values. This superior performance highlights the effectiveness of our BART-based architecture in capturing the complex relationships between molecular structures and biological activity.
Les modèles HBCVTr ont démontré des performances prédictives remarquables sur les ensembles de données VHB et VHC. Ils ont surpassé les approches conventionnelles d’apprentissage automatique, atteignant systématiquement des valeurs R carré et PCC plus élevées. Ces performances supérieures mettent en évidence l’efficacité de notre architecture basée sur BART pour capturer les relations complexes entre les structures moléculaires et l’activité biologique.
Virtual Screening and Pharmacokinetic Properties
Criblage virtuel et propriétés pharmacocinétiques
Virtual screening of 10 million compounds identified promising candidates with high predicted biological activity against HBV and HCV. These candidates exhibited favorable pharmacokinetic properties, including low molecular weight, good solubility, and low lipophilicity. Importantly, they demonstrated a low potential for pan-assay interference and structural alerts for potential toxicity.
Le criblage virtuel de 10 millions de composés a identifié des candidats prometteurs présentant une activité biologique prévue élevée contre le VHB et le VHC. Ces candidats présentaient des propriétés pharmacocinétiques favorables, notamment un faible poids moléculaire, une bonne solubilité et une faible lipophilie. Surtout, ils ont démontré un faible potentiel d’interférence avec les tests panoramiques et des alertes structurelles en cas de toxicité potentielle.
Molecular Docking and Molecular Dynamics Simulation
Docking moléculaire et simulation de dynamique moléculaire
Molecular docking and molecular dynamics simulations revealed the potential binding modes of the top candidates to target proteins. The complexes exhibited stable interactions, indicating the potential for these candidates to inhibit HBV and HCV. Further studies are warranted to validate their antiviral activity and elucidate their mechanisms of action.
Les simulations d’amarrage moléculaire et de dynamique moléculaire ont révélé les modes de liaison potentiels des meilleurs candidats aux protéines cibles. Les complexes ont présenté des interactions stables, indiquant le potentiel de ces candidats à inhiber le VHB et le VHC. D'autres études sont nécessaires pour valider leur activité antivirale et élucider leurs mécanismes d'action.
Discussion
Discussion
The HBCVTr virtual screening tool represents a significant advancement in drug discovery for HBV and HCV. Our AI-powered approach enables the rapid identification of potential inhibitors, significantly accelerating the process of developing new antiviral therapies. The integration of pharmacokinetic properties prediction and molecular docking/dynamics simulations provides valuable insights into the potential drug-like characteristics and binding mechanisms of the candidates.
L'outil de dépistage virtuel HBCVTr représente une avancée significative dans la découverte de médicaments contre le VHB et le VHC. Notre approche basée sur l'IA permet l'identification rapide d'inhibiteurs potentiels, accélérant ainsi considérablement le processus de développement de nouvelles thérapies antivirales. L’intégration de la prédiction des propriétés pharmacocinétiques et des simulations d’amarrage/dynamique moléculaire fournit des informations précieuses sur les caractéristiques potentielles des médicaments et les mécanismes de liaison des candidats.
The HBCVTr tool has broad implications for the field of drug discovery. It can be easily adapted to screen for inhibitors of other viruses, bacteria, and parasites, contributing to the development of personalized and targeted treatments for infectious diseases. Moreover, its underlying AI architecture can be leveraged to predict a wide range of biological activities, facilitating the discovery of novel drugs for various therapeutic applications.
L'outil HBCVTr a de vastes implications dans le domaine de la découverte de médicaments. Il peut être facilement adapté pour dépister les inhibiteurs d’autres virus, bactéries et parasites, contribuant ainsi au développement de traitements personnalisés et ciblés contre les maladies infectieuses. De plus, son architecture d’IA sous-jacente peut être exploitée pour prédire un large éventail d’activités biologiques, facilitant ainsi la découverte de nouveaux médicaments pour diverses applications thérapeutiques.
Conclusion
Conclusion
In conclusion, the HBCVTr virtual screening tool is a powerful AI-driven platform that transforms the drug discovery process for HBV and HCV. Its exceptional predictive performance, coupled with comprehensive pharmacokinetic and molecular docking/dynamics simulations, enables the rapid identification and characterization of promising antiviral candidates. As we continue to harness the transformative power of AI, we anticipate further advancements in drug discovery, leading to the development of effective and accessible treatments for a myriad of diseases.
En conclusion, l’outil de dépistage virtuel HBCVTr est une puissante plateforme basée sur l’IA qui transforme le processus de découverte de médicaments contre le VHB et le VHC. Ses performances prédictives exceptionnelles, associées à des simulations pharmacocinétiques et moléculaires/dynamiques complètes, permettent l’identification et la caractérisation rapides de candidats antiviraux prometteurs. Alors que nous continuons à exploiter le pouvoir transformateur de l’IA, nous prévoyons de nouveaux progrès dans la découverte de médicaments, conduisant au développement de traitements efficaces et accessibles pour une myriade de maladies.
Clause de non-responsabilité:info@kdj.com
Les informations fournies ne constituent pas des conseils commerciaux. kdj.com n’assume aucune responsabilité pour les investissements effectués sur la base des informations fournies dans cet article. Les crypto-monnaies sont très volatiles et il est fortement recommandé d’investir avec prudence après une recherche approfondie!
Si vous pensez que le contenu utilisé sur ce site Web porte atteinte à vos droits d’auteur, veuillez nous contacter immédiatement (info@kdj.com) et nous le supprimerons dans les plus brefs délais.
-

- Naviguer dans la tempête cryptographique : des Altcoins à l'épreuve de la récession à surveiller en 2025
- Oct 17, 2025 at 12:05 am
- Au milieu de la volatilité du marché, découvrez les meilleurs altcoins comme Digitap, Solana et Bittensor, positionnés pour prospérer en tant qu'investissements cryptographiques à l'épreuve de la récession en 2025.
-

- Binance, Bitcoin et Ethereum : naviguer dans les eaux agitées de la cryptographie
- Oct 17, 2025 at 12:02 am
- Les récentes turbulences du marché ont vu Binance se débarrasser de Bitcoin et d'Ethereum, suscitant des craintes de krach. De plus, la résurgence d'Ethereum L1 et l'amour Bitcoin de Steak 'n Shake.
-

- Eric Trump, Jeton immobilier et propriété : une nouvelle ère d’investissement ?
- Oct 17, 2025 at 12:00 am
- World Liberty Financial d'Eric Trump fait des vagues en symbolisant l'immobilier, démocratisant ainsi potentiellement l'accès aux propriétés haut de gamme. Est-ce l’avenir de l’investissement immobilier ?
-

- Or contre Bitcoin : confrontation avec la capitalisation boursière et ce que cela signifie pour vous
- Oct 17, 2025 at 12:00 am
- La capitalisation boursière croissante de l'or rivalise avec celle du Bitcoin, signalant un changement potentiel dans le sentiment des investisseurs et un possible retour vers la cryptographie.
-

-

- La dynastie crypto de la famille Trump : rendre à l'Amérique (et à ses portefeuilles) sa grandeur ?
- Oct 16, 2025 at 11:53 pm
- La famille Trump s’est mise à fond sur la cryptographie, amassant une fortune numérique d’un milliard de dollars. Est-ce du génie, de l’opportunisme ou simplement une autre aventure folle dans le monde des actifs numériques ?
-

-

-

- La capitalisation boursière de la cryptographie atteint des sommets sans précédent : qu'est-ce qui motive la montée en flèche des actifs numériques ?
- Oct 16, 2025 at 11:46 pm
- La capitalisation boursière des cryptomonnaies atteint de nouveaux sommets ! Découvrez les principaux facteurs, de l'adoption institutionnelle à la clarté réglementaire, qui propulsent les actifs numériques vers des sommets sans précédent.



























































