|
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
Nachrichtenartikel zu Kryptowährungen
Künstliche Intelligenz zur Bekämpfung von Hepatitis nutzen: Virtuelles Screening-Tool identifiziert ruhende Hemmstoffe
Apr 23, 2024 at 05:02 am
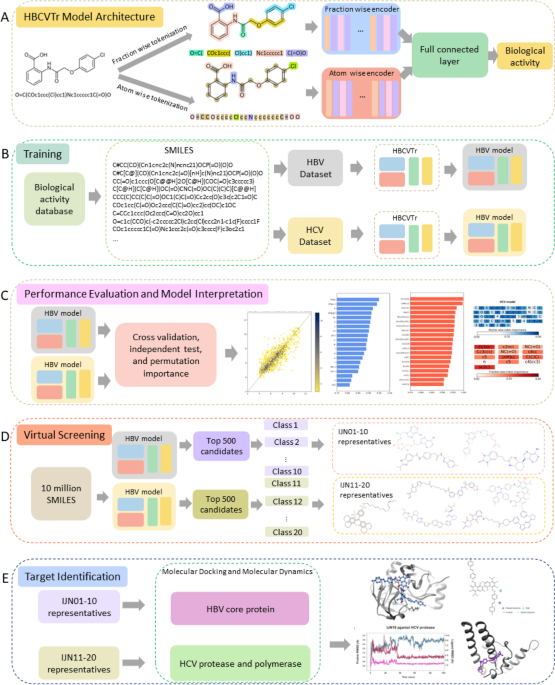
Eine neue Berechnungsmethode, HBCVTr, wurde entwickelt, um die antivirale Aktivität kleiner Moleküle gegen das Hepatitis-B-Virus (HBV) und das Hepatitis-C-Virus (HCV) anhand ihrer SMILES-Notationen vorherzusagen. Diese Methodik kombiniert eine bidirektionale und autoregressive Transformatorarchitektur (BART) mit atom- und bruchstückweisen Tokenisierungstechniken, um die sequentiellen Informationen und chemischen Merkmale von SMILES zu erfassen. Die Modelle wurden anhand kuratierter Datensätze mit 1941 bzw. 7454 Verbindungen für HBV und HCV trainiert und ausgewertet. Die HBCVTr-Modelle erzielten im Vergleich zu verschiedenen maschinellen Lernmodellen und bestehenden Methoden eine überlegene Leistung mit hoher Genauigkeit und robusten Vorhersagefähigkeiten. Virtuelles Screening mithilfe der HBCVTr-Modelle identifizierte potenzielle HBV- und HCV-Inhibitoren mit vielversprechenden pharmakokinetischen Eigenschaften. Molekulare Docking- und Molekulardynamiksimulationen validierten die Bindungsaffinität und Stabilität der Spitzenkandidaten weiter und lieferten Einblicke in ihre potenziellen Wirkmechanismen. Diese Methodik bietet ein wertvolles Werkzeug zur Entdeckung und Entwicklung neuartiger antiviraler Therapien gegen HBV und HCV.
Harnessing the Power of Artificial Intelligence for Drug Discovery: A Novel Virtual Screening Tool for Identifying Potential Inhibitors of Hepatitis B and C Viruses
Nutzung der Leistungsfähigkeit künstlicher Intelligenz für die Arzneimittelentwicklung: Ein neuartiges virtuelles Screening-Tool zur Identifizierung potenzieller Inhibitoren von Hepatitis-B- und -C-Viren
Abstract
Abstrakt
Hepatitis B and C viruses (HBV and HCV) pose significant global health challenges, necessitating the development of novel and effective antiviral therapies. To accelerate this process, we present HBCVTr, an innovative virtual screening tool that leverages artificial intelligence (AI) to identify potential inhibitors against these viruses. Our methodology incorporates a bidirectional and auto-regressive transformer (BART) architecture, trained on a vast dataset of SMILES notations and biological activity data. The HBCVTr model demonstrates exceptional predictive performance, surpassing conventional machine learning approaches. Through virtual screening of a library of 10 million compounds, we identified promising candidates with favorable pharmacokinetic properties. Molecular docking and dynamics simulations confirmed the potential of these candidates as inhibitors of HBV and HCV. Our findings underscore the transformative potential of AI in drug discovery, offering a rapid and efficient approach to identify novel therapeutic options for combating viral infections.
Hepatitis-B- und -C-Viren (HBV und HCV) stellen weltweit eine große Herausforderung für die Gesundheit dar und erfordern die Entwicklung neuartiger und wirksamer antiviraler Therapien. Um diesen Prozess zu beschleunigen, präsentieren wir HBCVTr, ein innovatives virtuelles Screening-Tool, das künstliche Intelligenz (KI) nutzt, um potenzielle Inhibitoren gegen diese Viren zu identifizieren. Unsere Methodik umfasst eine bidirektionale und autoregressive Transformer (BART)-Architektur, die auf einem umfangreichen Datensatz von SMILES-Notationen und Daten zur biologischen Aktivität trainiert wird. Das HBCVTr-Modell weist eine außergewöhnliche Vorhersageleistung auf und übertrifft herkömmliche Ansätze des maschinellen Lernens. Durch virtuelles Screening einer Bibliothek von 10 Millionen Verbindungen haben wir vielversprechende Kandidaten mit günstigen pharmakokinetischen Eigenschaften identifiziert. Molekulare Docking- und Dynamiksimulationen bestätigten das Potenzial dieser Kandidaten als Inhibitoren von HBV und HCV. Unsere Ergebnisse unterstreichen das transformative Potenzial von KI in der Arzneimittelforschung und bieten einen schnellen und effizienten Ansatz zur Identifizierung neuer Therapieoptionen zur Bekämpfung von Virusinfektionen.
Introduction
Einführung
Hepatitis B and C viruses are prevalent pathogens that infect millions worldwide, leading to severe liver damage and potential life-threatening complications. Despite the availability of antiviral therapies, the need for new and improved treatments remains urgent, particularly in the face of emerging drug resistance. Traditional drug discovery processes are often time-consuming and expensive, prompting the exploration of alternative approaches.
Hepatitis-B- und -C-Viren sind weit verbreitete Krankheitserreger, die weltweit Millionen Menschen infizieren und zu schweren Leberschäden und potenziell lebensbedrohlichen Komplikationen führen. Trotz der Verfügbarkeit antiviraler Therapien bleibt der Bedarf an neuen und verbesserten Behandlungen dringend, insbesondere angesichts der sich abzeichnenden Arzneimittelresistenz. Herkömmliche Verfahren zur Arzneimittelentdeckung sind oft zeitaufwändig und teuer und erfordern die Erforschung alternativer Ansätze.
In this study, we present HBCVTr, a groundbreaking virtual screening tool that harnesses the power of AI to expedite the identification of potential HBV and HCV inhibitors. Our methodology utilizes a BART architecture, renowned for its ability to process sequential data, to predict the biological activity of small molecules using SMILES notations. This approach enables the rapid screening of vast chemical libraries, significantly accelerating the drug discovery process.
In dieser Studie stellen wir HBCVTr vor, ein bahnbrechendes virtuelles Screening-Tool, das die Leistungsfähigkeit der KI nutzt, um die Identifizierung potenzieller HBV- und HCV-Inhibitoren zu beschleunigen. Unsere Methodik nutzt eine BART-Architektur, die für ihre Fähigkeit bekannt ist, sequentielle Daten zu verarbeiten, um die biologische Aktivität kleiner Moleküle mithilfe von SMILES-Notationen vorherzusagen. Dieser Ansatz ermöglicht das schnelle Screening umfangreicher chemischer Bibliotheken und beschleunigt so den Prozess der Arzneimittelentdeckung erheblich.
Methods
Methoden
Data Collection and Preprocessing
Datenerfassung und Vorverarbeitung
To train and evaluate our HBCVTr models, we curated antiviral activity assay data for HBV and HCV from the ChEMBL database. The data underwent rigorous filtering to ensure consistency and comparability, resulting in 1941 and 7454 compounds for HBV and HCV, respectively.
Um unsere HBCVTr-Modelle zu trainieren und zu bewerten, haben wir Daten zu antiviralen Aktivitätstests für HBV und HCV aus der ChEMBL-Datenbank zusammengestellt. Die Daten wurden einer strengen Filterung unterzogen, um Konsistenz und Vergleichbarkeit sicherzustellen. Das Ergebnis waren 1941 und 7454 Verbindungen für HBV bzw. HCV.
SMILES notations, representing the molecular structures of the compounds, were preprocessed to remove salts and convert them into canonical SMILES using the RDKit package. These SMILES were subsequently tokenized into atom-wise and fraction-wise tokens, capturing both individual atoms and unique functional groups.
SMILES-Notationen, die die molekularen Strukturen der Verbindungen darstellen, wurden vorverarbeitet, um Salze zu entfernen und sie mithilfe des RDKit-Pakets in kanonische SMILES umzuwandeln. Diese SMILES wurden anschließend in atom- und bruchstückweise Token umgewandelt, die sowohl einzelne Atome als auch einzigartige funktionelle Gruppen erfassten.
Model Architecture and Training
Modellarchitektur und Training
Our HBCVTr model is based on a BART architecture, which is specifically designed for sequential data processing. The model comprises two encoders: one for atom-wise tokens and the other for fraction-wise tokens. These encoders leverage multi-head attention layers to learn the contextual relationships between tokens. The outputs from the encoders are concatenated and passed through fully connected layers, culminating in a regression head that predicts the biological activity of the input SMILES.
Unser HBCVTr-Modell basiert auf einer BART-Architektur, die speziell für die sequentielle Datenverarbeitung konzipiert ist. Das Modell umfasst zwei Encoder: einen für atomweise Token und einen für bruchweise Token. Diese Encoder nutzen Multi-Head-Aufmerksamkeitsebenen, um die kontextuellen Beziehungen zwischen Token zu lernen. Die Ausgaben der Encoder werden verkettet und durch vollständig verbundene Schichten geleitet, was in einem Regressionskopf gipfelt, der die biologische Aktivität der eingegebenen SMILES vorhersagt.
We optimized the model's hyperparameters through a comprehensive grid search, ensuring optimal performance. The model was trained on 72% of the data, while 8% and 20% were allocated for validation and independent testing, respectively.
Wir haben die Hyperparameter des Modells durch eine umfassende Rastersuche optimiert und so eine optimale Leistung sichergestellt. Das Modell wurde auf 72 % der Daten trainiert, während 8 % bzw. 20 % für die Validierung bzw. unabhängige Tests vorgesehen waren.
Evaluation Criteria
Evaluationskriterien
To assess the predictive performance of HBCVTr, we employed a suite of regression evaluation metrics, including mean square error (MSE), mean absolute error (MAE), root mean square error (RMSE), R-squared, Pearson's correlation coefficient (PCC), and Spearman rank correlation (Spearman). These metrics evaluate the model's ability to accurately predict biological activity values.
Um die Vorhersageleistung von HBCVTr zu bewerten, verwendeten wir eine Reihe von Regressionsbewertungsmetriken, darunter mittlerer quadratischer Fehler (MSE), mittlerer absoluter Fehler (MAE), mittlerer quadratischer Fehler (RMSE), R-Quadrat und Pearson-Korrelationskoeffizient (PCC). und Spearman-Rangkorrelation (Spearman). Diese Metriken bewerten die Fähigkeit des Modells, biologische Aktivitätswerte genau vorherzusagen.
Virtual Screening and Pharmacokinetic Properties Prediction
Virtuelles Screening und Vorhersage pharmakokinetischer Eigenschaften
The trained HBCVTr models were utilized for virtual screening of a library of 10 million compounds. The top candidates with the highest predicted biological activity were further evaluated for their pharmacokinetic properties using the SwissADME web tool. This assessment ensured the identification of compounds with desirable drug-like characteristics.
Die trainierten HBCVTr-Modelle wurden für das virtuelle Screening einer Bibliothek von 10 Millionen Verbindungen verwendet. Die Spitzenkandidaten mit der höchsten vorhergesagten biologischen Aktivität wurden mithilfe des SwissADME-Webtools weiter auf ihre pharmakokinetischen Eigenschaften hin untersucht. Diese Bewertung stellte die Identifizierung von Verbindungen mit wünschenswerten arzneimittelähnlichen Eigenschaften sicher.
Molecular Docking and Molecular Dynamics Simulation
Molekulares Docking und Molekulardynamiksimulation
To investigate the potential binding of the top candidates to target proteins, molecular docking was performed using Autodock Vina. The stability of the protein-ligand complexes was subsequently assessed through molecular dynamics simulations using the Desmond Molecular Dynamics System. These simulations provided insights into the interactions between the candidates and their target proteins.
Um die potenzielle Bindung der Spitzenkandidaten an Zielproteine zu untersuchen, wurde molekulares Docking mit Autodock Vina durchgeführt. Die Stabilität der Protein-Ligand-Komplexe wurde anschließend durch Molekulardynamiksimulationen mit dem Desmond Molecular Dynamics System bewertet. Diese Simulationen lieferten Einblicke in die Wechselwirkungen zwischen den Kandidaten und ihren Zielproteinen.
Results
Ergebnisse
Model Performance
Modellleistung
The HBCVTr models demonstrated remarkable predictive performance on both HBV and HCV datasets. They outperformed conventional machine learning approaches, consistently achieving higher R-squared and PCC values. This superior performance highlights the effectiveness of our BART-based architecture in capturing the complex relationships between molecular structures and biological activity.
Die HBCVTr-Modelle zeigten eine bemerkenswerte Vorhersageleistung sowohl bei HBV- als auch bei HCV-Datensätzen. Sie übertrafen herkömmliche Ansätze des maschinellen Lernens und erzielten durchweg höhere R-Quadrat- und PCC-Werte. Diese überlegene Leistung unterstreicht die Wirksamkeit unserer BART-basierten Architektur bei der Erfassung der komplexen Beziehungen zwischen molekularen Strukturen und biologischer Aktivität.
Virtual Screening and Pharmacokinetic Properties
Virtuelles Screening und pharmakokinetische Eigenschaften
Virtual screening of 10 million compounds identified promising candidates with high predicted biological activity against HBV and HCV. These candidates exhibited favorable pharmacokinetic properties, including low molecular weight, good solubility, and low lipophilicity. Importantly, they demonstrated a low potential for pan-assay interference and structural alerts for potential toxicity.
Virtuelles Screening von 10 Millionen Verbindungen identifizierte vielversprechende Kandidaten mit hoher vorhergesagter biologischer Aktivität gegen HBV und HCV. Diese Kandidaten zeigten günstige pharmakokinetische Eigenschaften, einschließlich niedrigem Molekulargewicht, guter Löslichkeit und geringer Lipophilie. Wichtig ist, dass sie ein geringes Potenzial für Pan-Assay-Interferenzen und strukturelle Warnungen vor potenzieller Toxizität zeigten.
Molecular Docking and Molecular Dynamics Simulation
Molekulares Docking und Molekulardynamiksimulation
Molecular docking and molecular dynamics simulations revealed the potential binding modes of the top candidates to target proteins. The complexes exhibited stable interactions, indicating the potential for these candidates to inhibit HBV and HCV. Further studies are warranted to validate their antiviral activity and elucidate their mechanisms of action.
Molekulare Docking- und Molekulardynamiksimulationen enthüllten die möglichen Bindungsmodi der Spitzenkandidaten an Zielproteine. Die Komplexe zeigten stabile Wechselwirkungen, was auf das Potenzial dieser Kandidaten hindeutet, HBV und HCV zu hemmen. Weitere Studien sind erforderlich, um ihre antivirale Aktivität zu validieren und ihre Wirkmechanismen aufzuklären.
Discussion
Diskussion
The HBCVTr virtual screening tool represents a significant advancement in drug discovery for HBV and HCV. Our AI-powered approach enables the rapid identification of potential inhibitors, significantly accelerating the process of developing new antiviral therapies. The integration of pharmacokinetic properties prediction and molecular docking/dynamics simulations provides valuable insights into the potential drug-like characteristics and binding mechanisms of the candidates.
Das virtuelle Screening-Tool HBCVTr stellt einen bedeutenden Fortschritt in der Arzneimittelentwicklung für HBV und HCV dar. Unser KI-gestützter Ansatz ermöglicht die schnelle Identifizierung potenzieller Inhibitoren und beschleunigt so den Prozess der Entwicklung neuer antiviraler Therapien erheblich. Die Integration der Vorhersage pharmakokinetischer Eigenschaften und molekularer Docking-/Dynamiksimulationen liefert wertvolle Einblicke in die potenziellen wirkstoffähnlichen Eigenschaften und Bindungsmechanismen der Kandidaten.
The HBCVTr tool has broad implications for the field of drug discovery. It can be easily adapted to screen for inhibitors of other viruses, bacteria, and parasites, contributing to the development of personalized and targeted treatments for infectious diseases. Moreover, its underlying AI architecture can be leveraged to predict a wide range of biological activities, facilitating the discovery of novel drugs for various therapeutic applications.
Das HBCVTr-Tool hat weitreichende Auswirkungen auf den Bereich der Arzneimittelforschung. Es kann leicht angepasst werden, um nach Inhibitoren anderer Viren, Bakterien und Parasiten zu suchen und so zur Entwicklung personalisierter und gezielter Behandlungen für Infektionskrankheiten beizutragen. Darüber hinaus kann die zugrunde liegende KI-Architektur genutzt werden, um ein breites Spektrum biologischer Aktivitäten vorherzusagen und so die Entdeckung neuartiger Medikamente für verschiedene therapeutische Anwendungen zu erleichtern.
Conclusion
Abschluss
In conclusion, the HBCVTr virtual screening tool is a powerful AI-driven platform that transforms the drug discovery process for HBV and HCV. Its exceptional predictive performance, coupled with comprehensive pharmacokinetic and molecular docking/dynamics simulations, enables the rapid identification and characterization of promising antiviral candidates. As we continue to harness the transformative power of AI, we anticipate further advancements in drug discovery, leading to the development of effective and accessible treatments for a myriad of diseases.
Zusammenfassend lässt sich sagen, dass das virtuelle Screening-Tool HBCVTr eine leistungsstarke KI-gesteuerte Plattform ist, die den Arzneimittelentwicklungsprozess für HBV und HCV transformiert. Seine außergewöhnliche Vorhersageleistung, gepaart mit umfassenden pharmakokinetischen und molekularen Docking-/Dynamiksimulationen, ermöglicht die schnelle Identifizierung und Charakterisierung vielversprechender antiviraler Kandidaten. Während wir weiterhin die transformative Kraft der KI nutzen, erwarten wir weitere Fortschritte in der Arzneimittelforschung, die zur Entwicklung wirksamer und zugänglicher Behandlungen für eine Vielzahl von Krankheiten führen werden.
Haftungsausschluss:info@kdj.com
Die bereitgestellten Informationen stellen keine Handelsberatung dar. kdj.com übernimmt keine Verantwortung für Investitionen, die auf der Grundlage der in diesem Artikel bereitgestellten Informationen getätigt werden. Kryptowährungen sind sehr volatil und es wird dringend empfohlen, nach gründlicher Recherche mit Vorsicht zu investieren!
Wenn Sie glauben, dass der auf dieser Website verwendete Inhalt Ihr Urheberrecht verletzt, kontaktieren Sie uns bitte umgehend (info@kdj.com) und wir werden ihn umgehend löschen.
-

-

- Pepe hüpfte zu einer Marktkapitalisierung von 3,8 Mio. USD, aber seine Regierungszeit als Meme -Münz -Liebling verblasst. Geben Sie InfluencerPepe (Inpepe) ein
- Apr 04, 2025 at 03:15 am
- Inpepe ist ein Token, das nicht nur hier ist, um zu spielen - es ist hier, um mit echtem Nutzen zu dominieren. Wenn Sie bereit sind, von Pepes Froschfrosch-Vergangenheit weiterzumachen
-

- RWA Tokenization Platform Coldware (Cold) erstreckt sich 80% als SUI (SUI) und AVALANGE (AVAX)
- Apr 04, 2025 at 03:10 am
- In der sich ständig weiterentwickelnden Welt der Kryptowährung steigen einige Token zu Bedeutung, während andere wichtige Rückschläge erleben. In den jüngsten Entwicklungen fielen SUI (SUI) um 4,43%, und Avalanche (AVAX) kämpfte mit einem bemerkenswerten Rückgang von 13,83%.
-

- Top 3 Münzen, die in diesem April zu sehen sind: Hedera (HBAR), Binance Coin (BNB) und Kaltwaren (Kälte)
- Apr 04, 2025 at 03:10 am
- Beim Eintritt in den April 2025 zeigt der Kryptowährungsmarkt starke Signale für potenzielles Wachstum, wobei einige herausragende Münzen bereit sind, erhebliche Renditen zu erzielen.
-

-

-

- Trumps neues globales Tarif -Regime -Anlegerstimmung umgestaltet und löst scharfe Korrekturen über Risikovermögenswerte aus
- Apr 04, 2025 at 03:00 am
- Laut Marktanalysten verformt Präsident Donald Trumps neu angekündigtes globales Tarifregime bereits die Stimmung der Anleger und löst scharfe Korrekturen über Risikovermögen aus.
-

- Der Internal Revenue Service (IRS) verwendet die 1099-Form-Serie, um das Einkommen ohne Beschäftigung zu verfolgen, einschließlich Einnahmen aus Kryptowährungen.
- Apr 04, 2025 at 03:00 am
- Wenn eine Person durch Crypto -Plattformen Einkommen verdient, erhalten sie möglicherweise ein 1099 -Formular, das auch vom IRS von der Plattform vorgelegt wird.
-