
|
|

|
|

|
|

|
|

|
|

|
|

|
|

|
|

|
|

|
|

|
|

|
|

|
|

|
|

|
|
Nachrichtenartikel zu Kryptowährungen
MolE: Ein Transformatormodell für das Lernen molekularer Graphen
Nov 12, 2024 at 06:04 pm
Einführung von MolE, einem transformatorbasierten Modell für das Lernen molekularer Graphen. MolE arbeitet direkt mit molekularen Graphen, indem es sowohl Atomidentifikatoren als auch Graphkonnektivität als Eingabetoken bereitstellt. Atomidentifikatoren werden durch Hashing verschiedener Atomeigenschaften in einer einzigen Ganzzahl berechnet, und die Diagrammkonnektivität wird als topologische Distanzmatrix angegeben. MolE verwendet einen Transformer als Basisarchitektur, der bereits zuvor auch auf Diagramme angewendet wurde. Die Leistung von Transformatoren kann zu einem großen Teil auf die umfassende Nutzung des Selbstaufmerksamkeitsmechanismus zurückgeführt werden. In Standardtransformatoren werden die Eingabetokens in Abfragen, Schlüssel und Werte \(Q,K,V\in {R}^{N\times d}\) eingebettet, die zur Berechnung der Selbstaufmerksamkeit wie folgt verwendet werden:
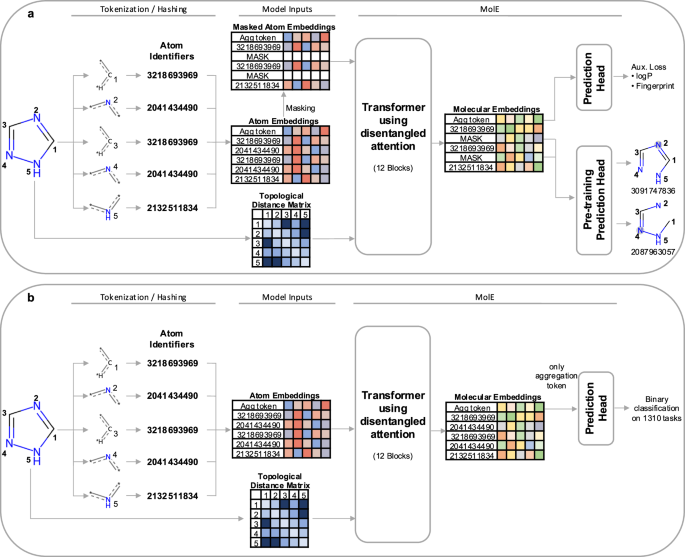
MolE is a transformer model designed specifically for molecular graphs. It directly works with graphs by providing both atom identifiers and graph connectivity as input tokens and relative position information, respectively. Atom identifiers are calculated by hashing different atomic properties into a single integer. In particular, this hash contains the following information:
MolE ist ein Transformatormodell, das speziell für molekulare Graphen entwickelt wurde. Es arbeitet direkt mit Graphen, indem es sowohl Atomkennungen als auch Graphkonnektivität als Eingabetoken bzw. relative Positionsinformationen bereitstellt. Atombezeichner werden berechnet, indem verschiedene Atomeigenschaften zu einer einzigen Ganzzahl gehasht werden. Insbesondere enthält dieser Hash die folgenden Informationen:
- number of neighboring heavy atoms,
- Anzahl benachbarter schwerer Atome,
- number of neighboring hydrogen atoms,
- Anzahl benachbarter Wasserstoffatome,
- valence minus the number of attached hydrogens,
- Wertigkeit minus Anzahl der gebundenen Wasserstoffe,
- atomic charge,
- Atomladung,
- atomic mass,
- Atommasse,
- attached bond types,
- angehängte Anleihetypen,
- and ring membership.
- und Ringmitgliedschaft.
Atom identifiers (also known as atom environments of radius 0) were computed using the Morgan algorithm as implemented in RDKit.
Atomidentifikatoren (auch bekannt als Atomumgebungen mit Radius 0) wurden mit dem Morgan-Algorithmus berechnet, wie er in RDKit implementiert ist.
In addition to tokens, MolE also takes graph connectivity information as input which is an important inductive bias since it encodes the relative position of atoms in the molecular graph. In this case, the graph connectivity is given as a topological distance matrix d where dij corresponds to the length of the shortest path over bonds separating atom i from atom j.
Neben Tokens verwendet MolE auch Informationen zur Graphkonnektivität als Eingabe, was eine wichtige induktive Vorspannung darstellt, da es die relative Position von Atomen im molekularen Graphen kodiert. In diesem Fall wird die Graphkonnektivität als topologische Distanzmatrix d angegeben, wobei dij der Länge des kürzesten Pfades über Bindungen entspricht, der Atom i von Atom j trennt.
MolE uses a Transformer as its base architecture, which also has been applied to graphs previously. The performance of transformers can be attributed in large part to the extensive use of the self-attention mechanism. In standard transformers, the input tokens are embedded into queries, keys and values \(Q,K,V\in {R}^{N\times d}\), which are used to compute self-attention as:
MolE verwendet einen Transformer als Basisarchitektur, der bereits zuvor auch auf Diagramme angewendet wurde. Die Leistung von Transformatoren kann zu einem großen Teil auf die umfassende Nutzung des Selbstaufmerksamkeitsmechanismus zurückgeführt werden. In Standardtransformatoren werden die Eingabetokens in Abfragen, Schlüssel und Werte \(Q,K,V\in {R}^{N\times d}\) eingebettet, die zur Berechnung der Selbstaufmerksamkeit wie folgt verwendet werden:
where \({H}_{0}\in {R}^{N\times d}\) are the output hidden vectors after self-attention, and \(d\) is the dimension of the hidden space.
wobei \({H}_{0}\in {R}^{N\times d}\) die ausgegebenen verborgenen Vektoren nach der Selbstaufmerksamkeit sind und \(d\) die Dimension des verborgenen Raums ist.
In order to explicitly carry positional information through each layer of the transformer, MolE uses the disentangled self-attention from DeBERTa:
Um Positionsinformationen explizit durch jede Schicht des Transformators zu transportieren, nutzt MolE die entwirrte Selbstaufmerksamkeit von DeBERTa:
where \({Q}^{c},{K}^{c},{V}^{c}\in {R}^{N\times d}\) are context queries, keys and values that contain token information (used in standard self-attention), and \({Q}_{i,j}^{p},{K}_{i,j}^{p}\in {R}^{N\times d}\) are the position queries and keys that encode the relative position of the \(i{{{\rm{th}}}}\) atom with respect to the \(j{{{\rm{th}}}}\) atom. The use of disentangled attention makes MolE invariant with respect to the order of the input atoms.
Dabei sind \({Q}^{c},{K}^{c},{V}^{c}\in {R}^{N\times d}\) Kontextabfragen, Schlüssel und Werte, die Token enthalten Informationen (verwendet in der Standard-Selbstaufmerksamkeit) und \({Q}_{i,j}^{p},{K}_{i,j}^{p}\in {R}^{N\times d}\) sind die Positionsabfragen und Schlüssel, die kodieren die relative Position des \(i{{{\rm{th}}}}\)-Atoms in Bezug auf das \(j{{{\rm{th}}}}\)-Atom. Durch die Verwendung der entwirrten Aufmerksamkeit wird MolE in Bezug auf die Reihenfolge der eingegebenen Atome invariant.
As mentioned earlier, self-supervised pretraining can effectively transfer information from large unlabeled datasets to smaller datasets with labels. Here we present a two-step pretraining strategy. The first step is a self-supervised approach to learn chemical structure representation. For this we use a BERT-like approach in which each atom is randomly masked with a probability of 15%, from which 80% of the selected tokens are replaced by a mask token, 10% replaced by a random token from the vocabulary, and 10% are not changed. Different from BERT, the prediction task is not to predict the identity of the masked token, but to predict the corresponding atom environment (or functional atom environment) of radius 2, meaning all atoms that are separated from the masked atom by two or less bonds. It is important to keep in mind that we used different tokenization strategies for inputs (radius 0) and labels (radius 2) and that input tokens do not contain overlapping data of neighboring atoms to avoid information leakage. This incentivizes the model to aggregate information from neighboring atoms while learning local molecular features. MolE learns via a classification task where each atom environment of radius 2 has a predefined label, contrary to the Context Prediction approach where the task is to match the embedding of atom environments of radius 4 to the embedding of context atoms (i.e., surrounding atoms beyond radius 4) via negative sampling. The second step uses a graph-level supervised pretraining with a large labeled dataset. As proposed by Hu et al., combining node- and graph-level pretraining helps to learn local and global features that improve the final prediction performance. More details regarding the pretraining steps can be found in the Methods section.
Wie bereits erwähnt, können durch selbstüberwachtes Vortraining Informationen effektiv von großen unbeschrifteten Datensätzen auf kleinere Datensätze mit Beschriftungen übertragen werden. Hier stellen wir eine zweistufige Pretraining-Strategie vor. Der erste Schritt ist ein selbstüberwachter Ansatz zum Erlernen der Darstellung chemischer Strukturen. Hierzu verwenden wir einen BERT-ähnlichen Ansatz, bei dem jedes Atom mit einer Wahrscheinlichkeit von 15 % zufällig maskiert wird, wobei 80 % der ausgewählten Token durch einen Masken-Token, 10 % durch einen zufälligen Token aus dem Vokabular usw. ersetzt werden 10 % werden nicht verändert. Anders als bei BERT besteht die Vorhersageaufgabe nicht darin, die Identität des maskierten Tokens vorherzusagen, sondern die entsprechende Atomumgebung (oder funktionale Atomumgebung) mit Radius 2 vorherzusagen, d. h. alle Atome, die durch zwei oder weniger Bindungen vom maskierten Atom getrennt sind . Es ist wichtig zu bedenken, dass wir unterschiedliche Tokenisierungsstrategien für Eingaben (Radius 0) und Beschriftungen (Radius 2) verwendet haben und dass Eingabetokens keine überlappenden Daten benachbarter Atome enthalten, um Informationslecks zu vermeiden. Dies gibt dem Modell einen Anreiz, Informationen von benachbarten Atomen zu aggregieren und gleichzeitig lokale molekulare Merkmale zu lernen. MolE lernt über eine Klassifizierungsaufgabe, bei der jede Atomumgebung mit Radius 2 eine vordefinierte Bezeichnung hat, im Gegensatz zum Kontextvorhersage-Ansatz, bei dem die Aufgabe darin besteht, die Einbettung von Atomumgebungen mit Radius 4 mit der Einbettung von Kontextatomen (d. h. umgebenden Atomen darüber hinaus) abzugleichen Radius 4) durch Negativabtastung. Im zweiten Schritt wird ein überwachtes Vortraining auf Diagrammebene mit einem großen beschrifteten Datensatz verwendet. Wie von Hu et al. vorgeschlagen, hilft die Kombination von Vortraining auf Knoten- und Diagrammebene beim Erlernen lokaler und globaler Merkmale, die die endgültige Vorhersageleistung verbessern. Weitere Einzelheiten zu den Vortrainingsschritten finden Sie im Abschnitt „Methoden“.
MolE was pretrained using an ultra-large database of ~842 million molecules from ZINC and ExCAPE-DB, employing a self-supervised scheme (with an auxiliary loss) followed by a supervised pretraining with ~456K molecules (see Methods section for more details). We assess the quality of the molecular embedding by finetuning MolE on a set of downstream tasks. In this case, we use a set of 22 ADMET tasks included in the Therapeutic Data Commons (TDC) benchmark This benchmark is composed of 9 regression and 13 binary classification tasks on datasets that range from hundreds (e.g, DILI with 475 compounds) to thousands of compounds (such as CYP inhibition tasks with ~13,000 compounds). An advantage of using this benchmark is
MolE wurde unter Verwendung einer extrem großen Datenbank mit ca. 842 Millionen Molekülen von ZINC und ExCAPE-DB vorab trainiert, wobei ein selbstüberwachtes Schema (mit einem Hilfsverlust) verwendet wurde, gefolgt von einem überwachten Vortraining mit ca. 456.000 Molekülen (weitere Einzelheiten finden Sie im Abschnitt „Methoden“). . Wir bewerten die Qualität der molekularen Einbettung, indem wir MolE auf eine Reihe nachgelagerter Aufgaben abstimmen. In diesem Fall verwenden wir einen Satz von 22 ADMET-Aufgaben, die im Therapeutic Data Commons (TDC)-Benchmark enthalten sind. Dieser Benchmark besteht aus 9 Regressions- und 13 binären Klassifizierungsaufgaben für Datensätze, die von Hunderten (z. B. DILI mit 475 Verbindungen) bis Tausenden reichen von Verbindungen (z. B. CYP-Inhibitionsaufgaben mit ~13.000 Verbindungen). Ein Vorteil der Verwendung dieses Benchmarks ist
Haftungsausschluss:info@kdj.com
Die bereitgestellten Informationen stellen keine Handelsberatung dar. kdj.com übernimmt keine Verantwortung für Investitionen, die auf der Grundlage der in diesem Artikel bereitgestellten Informationen getätigt werden. Kryptowährungen sind sehr volatil und es wird dringend empfohlen, nach gründlicher Recherche mit Vorsicht zu investieren!
Wenn Sie glauben, dass der auf dieser Website verwendete Inhalt Ihr Urheberrecht verletzt, kontaktieren Sie uns bitte umgehend (info@kdj.com) und wir werden ihn umgehend löschen.
-

-

- Shiba Inu (SHIB) Meme erkämpft sich die Top Ten
- Nov 14, 2024 at 10:25 pm
- Shiba Inu ist in den letzten 7 Tagen um erstaunliche 29 % gestiegen und hat 0,0000259 $ erreicht. Damit steigt die Marktkapitalisierung von Shiba Inu auf über 15,3 Milliarden US-Dollar und sichert ihm einen Platz im begehrten Top-10-Kryptoindex für die Marktkapitalisierung von Münzen. Dies könnte der Beginn eines wahnsinnigen Laufs für Shiba Inu sein, der mit der Stärke seiner Aufwärtsimpulse in der Vergangenheit immer wieder überraschende Aufwärtsbewegungen erlebte.
-

- Bitcoin erreicht Allzeithöchstpreis (ATH) von 1,47 Milliarden IDR, Analysten warnen vor kurzfristiger Preiskorrektur
- Nov 14, 2024 at 10:25 pm
- Am Mittwoch, gestern Abend um 11:20 Uhr WIB, erreichte Bitcoin seinen Allzeithöchstpreis (ATH) bei 93.434.000 US-Dollar oder umgerechnet 1,47 Milliarden Rupien pro Münze.
-

- Die Beerdigung des verstorbenen Schauspielers Song Jae-rim findet in düsterer Stimmung statt
- Nov 14, 2024 at 10:15 pm
- Die letzten Riten für die Beerdigung des verstorbenen Schauspielers Song Jae-rim fanden am 14. November um 12 Uhr im Yeouido St. Mary's Hospital im Seouler Bezirk Yeongdeungpo statt.
-

- Die TRON- und Avalanche-Preise sinken deutlich, was Anleger dazu veranlasst, ihre Anlageoptionen zu überdenken
- Nov 14, 2024 at 10:15 pm
- Während die meisten Münzen nach einer günstigen Wende der externen Effekte bullisch sind, waren TRX und AVAX das genaue Gegenteil. Die Preise von AVAX und Tron sind in den letzten Wochen deutlich gesunken, was Anleger dazu veranlasst, ihre Anlagemöglichkeiten zu überdenken.
-

-

- Der Preis von Bitcoin (BTC) strebt ein Ziel von über 100.000 US-Dollar an, da das bullische Cup-and-Handle-Muster auf einen möglichen Anstieg auf 255.000 US-Dollar hindeutet
- Nov 14, 2024 at 10:15 pm
- Analysten haben ein Ziel von über 100.000 US-Dollar für Bitcoin im Auge, wobei ein bullisches Cup-and-Handle-Muster auf einen möglichen Anstieg auf 255.000 US-Dollar hindeutet.
-

- BlackRock führt neue BUIDL-Anteilsklassen über mehrere Blockchains hinweg ein
- Nov 14, 2024 at 10:15 pm
- Wir freuen uns, Ihnen mitteilen zu können, dass der von Securitize tokenisierte @BlackRock USD Institutional Digital Liquidity Fund auf @Aptos, @Arbitrum, @Avax, @Optimism und @0xPolygon expandiert. Diese Mehrkette
-

- Die verborgenen Auswirkungen von Dogecoin: Revolutionierung von Industrien und Community-Dynamik
- Nov 14, 2024 at 10:15 pm
- Dogecoin, einst als bloßes Internet-Meme abgetan, hat nun seinen Status als herausragender Akteur im Bereich der Kryptowährungen gefestigt. Obwohl sein Aufstieg ausführlich diskutiert wurde, haben einige Aspekte nicht die Aufmerksamkeit erhalten, die sie verdienen. In diesem Artikel befassen wir uns mit unerforschten Aspekten von Dogecoin, die das Leben weltweit erheblich beeinflussen könnten.






























