 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
 |
|
Nachrichtenartikel zu Kryptowährungen
METAGENE-1: A Metagenomic Foundation Model for Biosurveillance and Pandemic Preparedness
Jan 07, 2025 at 10:51 am
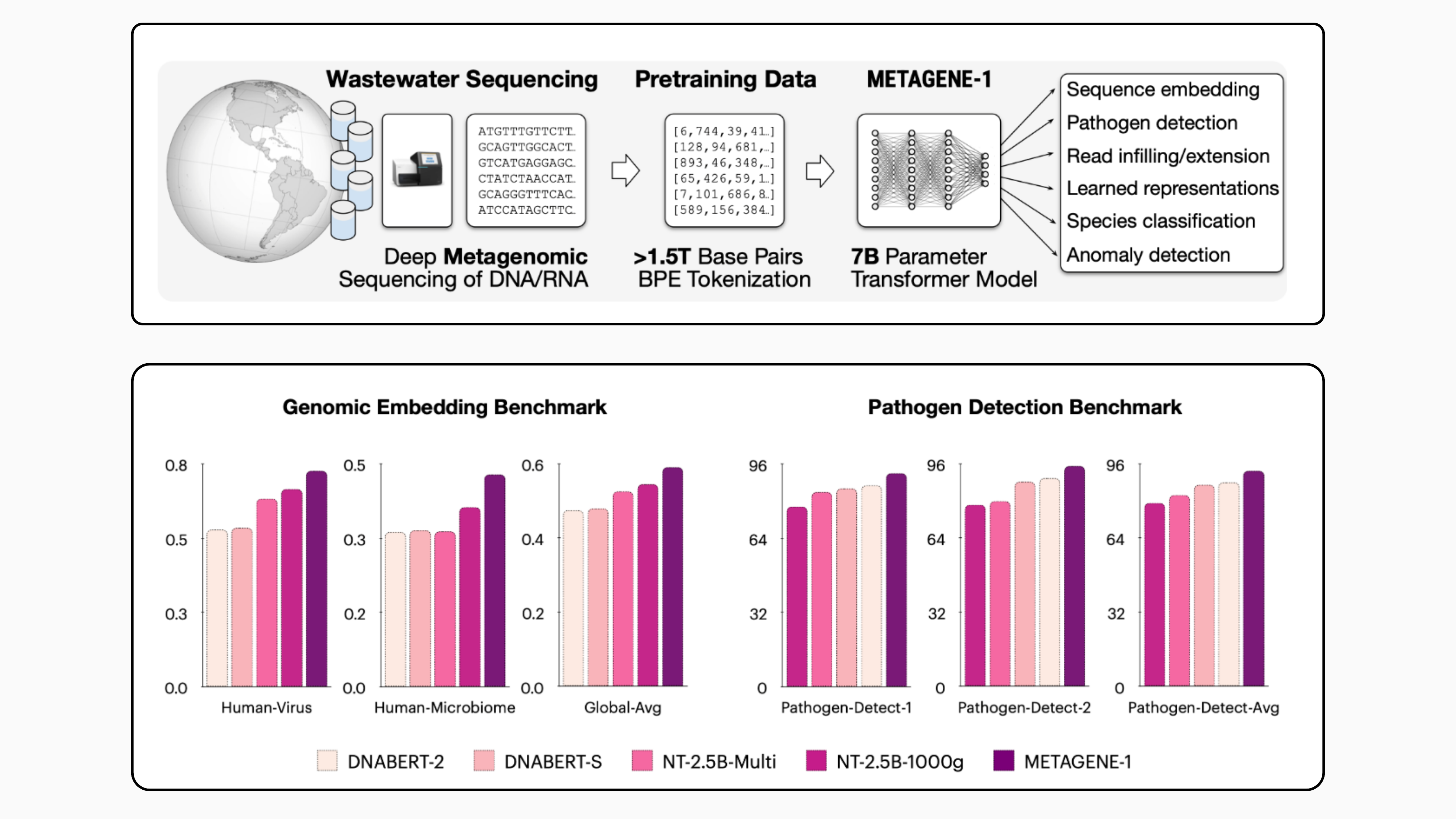
With emerging pandemics posing persistent threats to global health, the need for advanced biosurveillance and pathogen detection systems is becoming increasingly evident. Traditional genomic analysis methods, while effective in isolated cases, often encounter challenges in addressing the complexities of large-scale health monitoring. A significant difficulty lies in identifying and understanding the genomic diversity in environments such as wastewater, which contains a rich mix of microbial and viral DNA and RNA. In this context, the rapid advancements in biological research are highlighting the importance of scalable, accurate, and interpretable models to analyze vast amounts of metagenomic data, aiding in the prediction and mitigation of health crises.
Now, a team of researchers from the University of Southern California, Prime Intellect, and the Nucleic Acid Observatory have introduced METAGENE-1, a metagenomic foundation model. This 7-billion-parameter autoregressive transformer model is specifically designed to analyze metagenomic sequences. METAGENE-1 is trained on a dataset comprising over 1.5 trillion DNA and RNA base pairs derived from human wastewater samples, utilizing next-generation sequencing technologies and a tailored byte-pair encoding (BPE) tokenization strategy to capture the intricate genomic diversity present in these datasets. The model is open-sourced, encouraging collaboration and further advancements in the field.
Technical Highlights and BenefitsMETAGENE-1’s architecture draws on modern transformer models, including GPT and Llama families. This decoder-only transformer uses a causal language modeling objective to predict the next token in a sequence based on preceding tokens. Its key features include:
A decoder-only transformer architecture with 7 billion parameters.
Trained on a vast dataset of over 1.5 trillion DNA and RNA base pairs from human wastewater samples.
Employs a BPE tokenization strategy tailored to metagenomic sequences.
These features enable METAGENE-1 to generate high-quality sequence embeddings and adapt to specific tasks, enhancing its utility in the genomic and public health domains.
Results and InsightsThe capabilities of METAGENE-1 were assessed using multiple benchmarks, where it demonstrated notable performance. In a pathogen detection benchmark based on human wastewater samples, the model achieved an average Matthews correlation coefficient (MCC) of 92.96, significantly outperforming other models. Additionally, METAGENE-1 showed strong results in anomaly detection tasks, effectively distinguishing metagenomic sequences from other genomic data sources.
In embedding-based genomic analyses, METAGENE-1 excelled on the Gene-MTEB benchmark, achieving a global average score of 0.59. This performance underscores its adaptability in both zero-shot and fine-tuning scenarios, reinforcing its value in handling complex and diverse metagenomic data.
ConclusionMETAGENE-1 represents a thoughtful integration of artificial intelligence and metagenomics. By leveraging transformer architectures, the model offers practical solutions for biosurveillance and pandemic preparedness. Its open-source release invites researchers to collaborate and innovate, advancing the field of genomic science. As challenges related to emerging pathogens and global pandemics continue, METAGENE-1 demonstrates how technology can play a crucial role in addressing public health concerns effectively and responsibly.
Check out the Paper, Website, GitHub Page, and Model on Hugging Face. All credit for this research goes to the researchers of this project. Also, don’t forget to follow us on Twitter and join our Telegram Channel and LinkedIn Group. Don’t Forget to join our 60k+ ML SubReddit.
FREE UPCOMING AI WEBINAR (JAN 15, 2025): Boost LLM Accuracy with Synthetic Data and Evaluation Intelligence
Haftungsausschluss:info@kdj.com
Die bereitgestellten Informationen stellen keine Handelsberatung dar. kdj.com übernimmt keine Verantwortung für Investitionen, die auf der Grundlage der in diesem Artikel bereitgestellten Informationen getätigt werden. Kryptowährungen sind sehr volatil und es wird dringend empfohlen, nach gründlicher Recherche mit Vorsicht zu investieren!
Wenn Sie glauben, dass der auf dieser Website verwendete Inhalt Ihr Urheberrecht verletzt, kontaktieren Sie uns bitte umgehend (info@kdj.com) und wir werden ihn umgehend löschen.
-

-

- Qwen3Guard: Ein Sprung in Richtung mehrsprachiger KI -Sicherheit
- Sep 27, 2025 at 01:04 pm
- Entdecken Sie Alibabas QWEN3Guard, eine bahnbrechende mehrsprachige Sicherheitsmodellfamilie, die für die Echtzeit-Moderation von KI-Eingaben und -Antaktionen und die kalifornischen KI-Sicherheitsgesetze entwickelt wurde.
-

-

-

-

-

- Die Verbrennungsrate und Krypto -Gehaltsabrechnung von Shiba Inu: Eine New Yorker Perspektive
- Sep 27, 2025 at 11:01 am
- Entdecken Sie, wie Shiba Inus Verbrennungsrate Krypto -Gehaltsabrechnungen inspiriert. Tauchen Sie in das Engagement der Gemeinschaft, die finanzielle Inklusion und die Zukunft von Krypto ein.
-

-































































