Das Entwerfen synthetischer Reaktionswege für Moleküle ist ein grundlegender Aspekt der organischen Synthese und hat erhebliche Auswirkungen auf verschiedene Bereiche wie die biomedizinische, pharmazeutische und Materialindustrie. Die retrosynthetische Analyse ist der am weitesten verbreitete Ansatz zur Entwicklung synthetischer Routen.

Organic synthesis plays a pivotal role in various fields, including biomedical, pharmaceutical, and materials industries. Retrosynthetic analysis serves as the primary approach for designing synthetic routes, aiming to decompose molecules into simpler precursors using established reactions. This methodology, initially formalized by Corey, led to the development of computer-aided synthesis planning (CASP). In recent years, artificial intelligence (AI)-driven retrosynthesis has facilitated the exploration of more complex molecules and significantly reduced the time and energy required to design synthetic experiments. Single-step retrosynthesis prediction is a crucial component of retrosynthetic planning, and several deep learning-based methods have been proposed with promising results. These methods can be broadly categorized into three groups: template-based, template-free, and semi-template-based methods.
Die organische Synthese spielt in verschiedenen Bereichen eine zentrale Rolle, darunter in der biomedizinischen, pharmazeutischen und Materialindustrie. Die retrosynthetische Analyse dient als primärer Ansatz für die Entwicklung synthetischer Routen und zielt darauf ab, Moleküle mithilfe etablierter Reaktionen in einfachere Vorläufer zu zerlegen. Diese ursprünglich von Corey formalisierte Methodik führte zur Entwicklung der computergestützten Syntheseplanung (CASP). In den letzten Jahren hat die durch künstliche Intelligenz (KI) gesteuerte Retrosynthese die Erforschung komplexerer Moleküle erleichtert und den Zeit- und Energieaufwand für die Entwicklung synthetischer Experimente erheblich reduziert. Die Vorhersage der Retrosynthese in einem Schritt ist ein entscheidender Bestandteil der Retrosyntheseplanung, und es wurden mehrere Deep-Learning-basierte Methoden mit vielversprechenden Ergebnissen vorgeschlagen. Diese Methoden können grob in drei Gruppen eingeteilt werden: vorlagenbasierte, vorlagenfreie und halbvorlagenbasierte Methoden.
Template-based methods regard retrosynthesis prediction as a template retrieval problem and compare the target molecule with precomputed templates. These templates capture the essential features of the reaction center in specific types of chemical reactions. They can be generated manually or automatically and serve as a guide for the model to identify the most suitable chemical transformation for a given molecule. Various works have proposed different approaches to prioritize candidate templates. RetroSim employed the molecular fingerprint similarity between the given product and the molecules present in the corpus to rank the candidate templates. NeuralSym was the pioneering work to utilize deep neural networks for template selection by learning a multi-class classifier. GLN built a conditional graph logic network to learn the conditional joint probability of templates and reactants. LocalRetro conducted an evaluation of the suitability of local atom/bond templates at all predicted reaction centers for a target molecule and incorporated the non-local effects in chemical reactions through global reactivity attention. It has demonstrated state-of-the-art performance within the template-based methods. Although providing interpretability and molecule validity, template-based models suffer from limited generalization and scalability issues, which can hinder their practical utility.
Template-basierte Methoden betrachten die Retrosynthesevorhersage als ein Template-Retrieval-Problem und vergleichen das Zielmolekül mit vorberechneten Templates. Diese Vorlagen erfassen die wesentlichen Merkmale des Reaktionszentrums bei bestimmten Arten chemischer Reaktionen. Sie können manuell oder automatisch generiert werden und dienen dem Modell als Leitfaden zur Ermittlung der am besten geeigneten chemischen Umwandlung für ein bestimmtes Molekül. In verschiedenen Arbeiten wurden unterschiedliche Ansätze zur Priorisierung von Kandidatenvorlagen vorgeschlagen. RetroSim nutzte die Ähnlichkeit des molekularen Fingerabdrucks zwischen dem gegebenen Produkt und den im Korpus vorhandenen Molekülen, um die Kandidaten-Templates einzustufen. NeuralSym war die bahnbrechende Arbeit zur Nutzung tiefer neuronaler Netze für die Vorlagenauswahl durch das Erlernen eines Mehrklassen-Klassifikators. GLN baute ein bedingtes Diagrammlogiknetzwerk auf, um die bedingte gemeinsame Wahrscheinlichkeit von Vorlagen und Reaktanten zu lernen. LocalRetro führte eine Bewertung der Eignung lokaler Atom-/Bindungsvorlagen an allen vorhergesagten Reaktionszentren für ein Zielmolekül durch und berücksichtigte die nicht-lokalen Effekte in chemischen Reaktionen durch globale Reaktivitätsberücksichtigung. Es hat die Leistung der vorlagenbasierten Methoden auf dem neuesten Stand der Technik bewiesen. Obwohl vorlagenbasierte Modelle Interpretierbarkeit und Molekülvalidität bieten, weisen sie nur begrenzte Generalisierungs- und Skalierbarkeitsprobleme auf, die ihren praktischen Nutzen beeinträchtigen können.
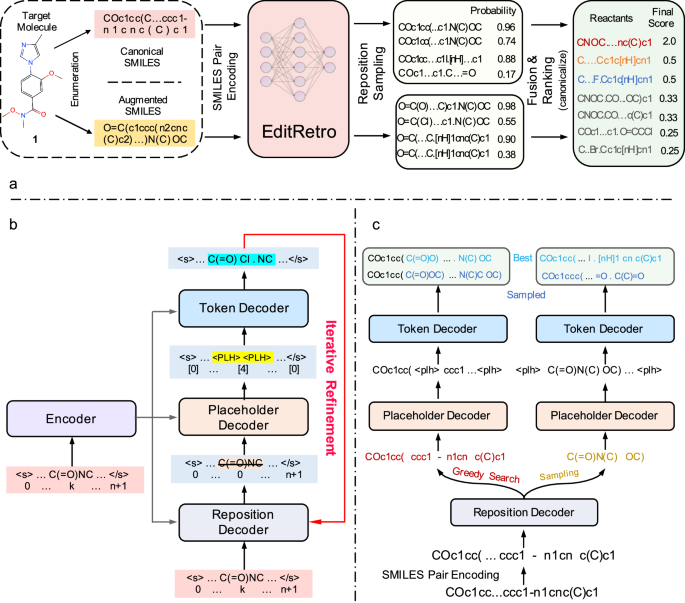
Template-free methods utilize deep generative models to generate reactant molecules without relying on predefined templates. Most of existing methods reformulate the task as a sequence-to-sequence problem, employing the sequence representation of molecules, specifically the simplified molecular-input line-entry system (SMILES). Liu et al. first utilized a long short-term memory (LSTM)-based sequence-to-sequence (Seq2Seq) model to convert the SMILES representation of a product to the SMILES of the reactants. Karpov et al. further proposed a Transformer-based Seq2Seq method for retrosynthesis. SCROP integrated a grammar corrector into the Transformer architecture, aiming to resolve the prevalent problem of grammatical invalidity in seq2seq methods. R-SMILES established a closely aligned one-to-one mapping between the SMILES representations of the products and the reactants to enhance the efficiency of synthesis prediction in Transformer-based methods. PMSR devised three tailored pre-training tasks for retrosynthesis, encompassing auto-regression, molecule recovery, and contrastive reaction classification, thereby enhancing the performance of retrosynthesis and achieving state-of-the-art accuracy within template-free methods. Some studies characterize the task as a graph-to-sequence problem, employing the molecular graph as input. Graph2SMILES integrated a sequential graph encoder with a Transformer decoder to preserve the permutation invariance of SMILES. Retroformer introduced a local attention head in the Transformer encoder to augment its reasoning capability for reactions. Recent studies, including MEGAN, MARS, and Graph2Edits, have explored the utilization of end-to-end molecular graph editing models to represent a chemical reaction as a series of graph edits, drawing inspiration from the arrow pushing formalism. However, these approaches usually require time-consuming predictions for sequential graph edit operations. Fang et al. developed a substructure-level decoding method by automatically extracting commonly preserved portions of product molecules. However, the extraction of substructures is fully data-driven, and its coverage depends on the reaction dataset. Furthermore, incorrect substructures can lead to erroneous predictions. While template-free methods are fully data-driven, they raise concerns regarding the interpretability, chemical validity, and diversity of the generated molecules.
Vorlagenfreie Methoden nutzen tiefe generative Modelle, um Reaktantenmoleküle zu erzeugen, ohne auf vordefinierte Vorlagen angewiesen zu sein. Die meisten vorhandenen Methoden formulieren die Aufgabe als Sequenz-zu-Sequenz-Problem um und verwenden dabei die Sequenzdarstellung von Molekülen, insbesondere das vereinfachte Molekular-Input-Line-Entry-System (SMILES). Liu et al. verwendeten zunächst ein auf dem langen Kurzzeitgedächtnis (LSTM) basierendes Sequenz-zu-Sequenz-Modell (Seq2Seq), um die SMILES-Darstellung eines Produkts in die SMILES-Darstellung der Reaktanten umzuwandeln. Karpov et al. schlug außerdem eine Transformer-basierte Seq2Seq-Methode für die Retrosynthese vor. SCROP hat einen Grammatikkorrektor in die Transformer-Architektur integriert, um das vorherrschende Problem der grammatikalischen Ungültigkeit in seq2seq-Methoden zu lösen. R-SMILES erstellte eine eng abgestimmte Eins-zu-Eins-Zuordnung zwischen den SMILES-Darstellungen der Produkte und der Reaktanten, um die Effizienz der Synthesevorhersage in Transformer-basierten Methoden zu verbessern. PMSR entwickelte drei maßgeschneiderte Vortrainingsaufgaben für die Retrosynthese, die Autoregression, Molekülwiederherstellung und Kontrastreaktionsklassifizierung umfassen, wodurch die Leistung der Retrosynthese verbessert und modernste Genauigkeit bei templatfreien Methoden erreicht wurde. Einige Studien charakterisieren die Aufgabe als ein Graph-zu-Sequenz-Problem, bei dem der molekulare Graph als Eingabe verwendet wird. Graph2SMILES hat einen sequentiellen Graph-Encoder mit einem Transformer-Decoder integriert, um die Permutationsinvarianz von SMILES zu bewahren. Retroformer hat im Transformer-Encoder einen lokalen Aufmerksamkeitskopf eingeführt, um dessen Argumentationsfähigkeit für Reaktionen zu verbessern. Aktuelle Studien, darunter MEGAN, MARS und Graph2Edits, haben die Verwendung von End-to-End-Modellen zur Bearbeitung molekularer Graphen untersucht, um eine chemische Reaktion als eine Reihe von Graphbearbeitungen darzustellen, wobei sie sich vom pfeilschiebenden Formalismus inspirieren ließen. Diese Ansätze erfordern jedoch normalerweise zeitaufwändige Vorhersagen für sequentielle Diagrammbearbeitungsvorgänge. Fang et al. entwickelte eine Dekodierungsmethode auf Unterstrukturebene, bei der häufig konservierte Teile von Produktmolekülen automatisch extrahiert wurden. Die Extraktion von Unterstrukturen ist jedoch vollständig datengesteuert und ihre Abdeckung hängt vom Reaktionsdatensatz ab. Darüber hinaus können falsche Unterstrukturen zu fehlerhaften Vorhersagen führen. Während templatfreie Methoden vollständig datengesteuert sind, werfen sie Bedenken hinsichtlich der Interpretierbarkeit, chemischen Gültigkeit und Diversität der erzeugten Moleküle auf.
Semi-template-based methods leverage the benefits of the two aforementioned methods. These methods follow a two-stage procedure: first, fragmenting the target molecule into synthons by identifying reactive sites, and subsequently converting the synthons into reactants using techniques such as leaving groups selection, graph generation, or SMILES generation. RetroXpert first identified the reaction center of the target molecule to obtain synthons by employing an edge-enhanced graph attention network, followed by the generation of the corresponding reactants based on the synthons. RetroPrime introduced the mix-and-match and label-and-align strategies within a Transformer-based two-stage workflow to mitigate the challenges of insufficient diversity and chemical implausibility. G2Gs initially partitioned the target molecular graph into several synthons by identifying potential reaction centers, followed by the translation of the synthons into the complete reactant graphs using a variational graph translation framework. GraphRetro first transformed the target into synthons by
Halbvorlagenbasierte Methoden nutzen die Vorteile der beiden oben genannten Methoden. Diese Methoden folgen einem zweistufigen Verfahren: Zunächst wird das Zielmolekül durch Identifizierung reaktiver Stellen in Synthone fragmentiert und anschließend werden die Synthone mithilfe von Techniken wie der Auswahl von Abgangsgruppen, der Erstellung von Graphen oder der SMILES-Erstellung in Reaktanten umgewandelt. RetroXpert identifizierte zunächst das Reaktionszentrum des Zielmoleküls, um Synthone zu erhalten, indem es ein kantenverstärktes Graph-Aufmerksamkeitsnetzwerk einsetzte, gefolgt von der Erzeugung der entsprechenden Reaktanten auf Basis der Synthone. RetroPrime führte die Mix-and-Match- und Label-and-Alignment-Strategien innerhalb eines Transformer-basierten zweistufigen Workflows ein, um die Herausforderungen unzureichender Diversität und chemischer Unplausibilität zu mindern. G2Gs teilten den Zielmolekülgraphen zunächst in mehrere Synthone auf, indem sie potenzielle Reaktionszentren identifizierten, gefolgt von der Übersetzung der Synthone in die vollständigen Reaktantengraphen mithilfe eines Variationsgraphenübersetzungsrahmens. GraphRetro wandelte das Ziel zunächst in Synthons um